Introducción
En los últimos años se ha discutido acerca del componente genético que puede definir a un organismo de manera individual. Al respecto, se podría pensar que, realizando un análisis del genoma de cada una de sus células, sería posible conocer la información necesaria que define su componente genético, sin embargo, incluso entre las células de un mismo individuo, se encontrarían muchas diferencias. Estas diferencias están dadas por mutaciones, recombinaciones y por otros factores que incluyen el contacto con otros organismos. Así, dentro de la comunidad científica se debate la premisa de que la naturaleza en sí, se encuentra conformada por miles de consorcios de múltiples especies, y que dichos consorcios están compuestos por un organismo hospedero y su microbioma (holobionte), en donde el consorcio podría estar actuando como una sola unidad de selección. Entonces, estas unidades englobarían animales y plantas, así como el conjunto de microorganismos que conforman su microbiota, y la suma de toda la información genética de los holobiontes, constituiría el hologenoma (Fig. 1).
Figura 1
Interacciones entre los holobiontes en la relación hospedero-vector de transmisión-microbioma
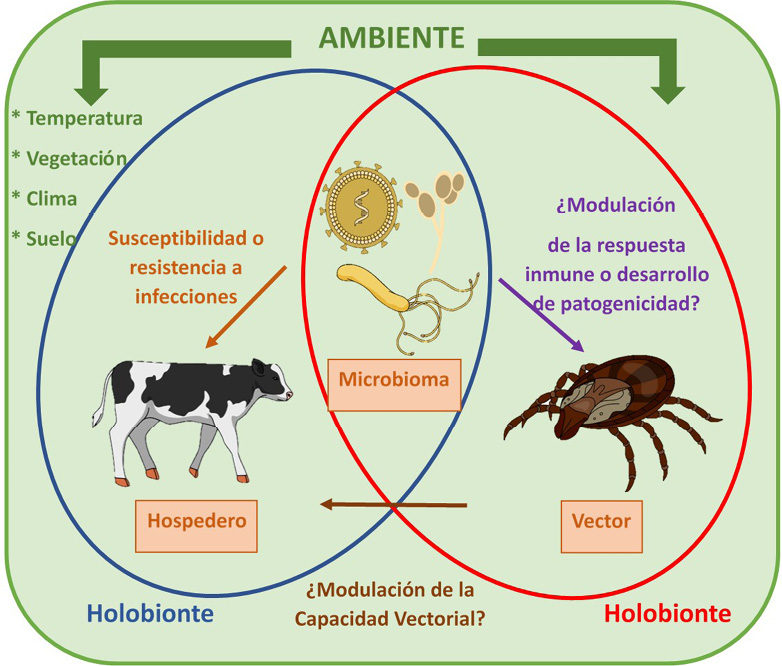
El hospedero y el vector de transmisión establecen una relación con su microbioma, el cual influye en diversos procesos moleculares y fisiológicos de los mismos. En este escenario, se contempla la influencia que ejerce en el ambiente, que también estaría modificando el microbioma.
Si se pudiera tan sólo pensar en el conjunto de bacterias que conviven con todos los organismos, se tendría una idea de lo complejo que podría resultar esta interacción. De esta forma, las bacterias que viven en el cuerpo de un ser vivo (microbioma), pueden influir de manera directa e indirecta en los diversos aspectos biológicos contribuyendo a la homeostasis del individuo. Por tal motivo, para evaluar la adecuación de un organismo, se debe de considerar no sólo su genoma, sino también los genomas de los organismos asociados (hologenoma).
Al igual que la gran diversidad que existen entre las bacterias, los artrópodos son el grupo de animales más megadiversos en la tierra, encontrándose prácticamente en todos los ecosistemas (Hills et al., 2005). Para subsistir, los artrópodos son capaces de aprovechar diversas fuentes de alimento, esto en gran medida, debido a la presencia de diversos microorganismos que les permiten obtener y metabolizar nutrientes (Hills et al., 2005). Además, muchas de estas interacciones artrópodo-microorganismos se encuentran relacionadas con diversos mecanismos e incluso, hasta con el desarrollo de órganos especializados que coadyuvan para regular el desarrollo de su microbiota. Por ejemplo, muchas de las bacterias que forman parte del microbioma en los artrópodos, generan biopelículas que son necesarias para el establecimiento de sus relaciones simbióticas en los holobiontes (Maltz et al., 2012; Kim et al., 2014; Purdy et al., 2011). Aunado a estos procesos, los artrópodos cuentan con un sistema inmunoendócrino que los protege de la mayoría de los patógenos, no obstante, los mecanismos de este sistema que están involucrados en la tolerancia ante los endosimbiontes y los patógenos que transmite, aún permanece sin esclarecerse. Así, muchos artrópodos funcionan biológicamente como vectores de una gran variedad de patógenos, como es el caso de las garrapatas, agentes causales de importantes enfermedades (Aguilar-Díaz et al., 2018).
Solamente por debajo de los mosquitos, las garrapatas ocupan el segundo lugar dentro de los artrópodos que trasmiten enfermedades a un gran número de hospederos vertebrados. Entre las diferentes garrapatas, la especie Rhipicephalus microplus, es el ectoparásito hematófago de ganado vacuno que causa más pérdidas económicas mundiales en la industria pecuaria. Esto se debe a su capacidad parasitaria y expoliativa, y a la transmisión vectorial de agentes infecciosos que causan enfermedades como la anaplasmosis y la babesiosis (George, 2000). Hasta 2013, el reporte del censo ganadero, estima que en México la población de ganado vacuno era de 32,402,461 individuos (SIAP-SAGARPA) y que el 75% de este ganado comparte zonas endémicas con la garrapata, lo que brinda un panorama de la magnitud de pérdidas económicas causadas por este parásito.
Actualmente, el control de R. microplus, se basa principalmente en el tratamiento del ganado con agentes acaricidas químicos (ixodicidas) en áreas endémicas de forma preventiva. Sin embargo, el uso excesivo de estos, ha favorecido la selección de resistencia de las garrapatas a estos compuestos (Abbas et al., 2014).
Por su condición de hematofagia, las garrapatas durante el proceso de ingurgitación son capaces de transmitir, o bien de adquirir, un gran número de microorganismos tanto patógenos como no-patógenos (Goddard et al., 2000; Hill et al., 2005). Además, las garrapatas son capaces de albergar una microbiota diversa conformada por bacterias, protozoarios, hongos y virus, que desempeñan funciones fundamentales en el establecimiento, reproducción, fisiología, nutrición, y probablemente también en la capacidad vectorial (Andreotti et al., 2011; Boissière et al., 2012; Broderick et al., 2012; Dillon et al., 2004). Las comunidades microbianas de la garrapata, se han relacionado con la capacidad que tienen algunos patógenos para colonizar y sobrevivir dentro del vector, así como la habilidad de transmitirse de manera eficaz a su hospedero (Cirimotich et al., 2011; Narasimhan et al., 2014; Weiss 2011). En este sentido, a pesar de los avances en la investigación sobre la biología de garrapatas, el estudio del microbioma representa una excelente alternativa para la elucidación del papel que tienen las comunidades microbianas en la interacción con los patógenos, la fisiología y la capacidad vectorial sobre su hospedero. El comprender dichas interacciones entre el microbioma de los artrópodos y los patógenos que transmite, abre la posibilidad de proporcionar nuevas estrategias para controlar y prevenir las enfermedades transmitidas por vectores.
1. Estudio del microbioma en garrapatas
La investigación del microbioma ha ganado gran proyección, luego de descubrir su participación en el cáncer, las enfermedades neurodegenerativas, los trastornos metabólicos, la inmunidad y más recientemente el envejecimiento (Swei et al., 2017; Graey et al., 2018). El estudio del papel del microbioma se ha involucrado en varios aspectos relacionados con el proceso salud-enfermedad, y la relación hospedero-patógeno, además, de destacar el estudio del microbioma de los vectores. Dentro de estos procesos, se incluyen la biosíntesis de vitaminas y aminoácidos, la desintoxicación xenobiótica, la regulación del sistema inmune del hospedero y del vector, la resistencia a drogas y su papel en la capacidad de trasmisión de patógenos. Recientemente, el papel del microbioma de las garrapatas en la transmisión de enfermedades infecciosas y de los mecanismos moleculares asociados, han recibido mucha atención. Se sabe que estos ectoparásitos hematófagos poseen una gran capacidad vectorial para transmitir una enorme variedad de patógenos causantes de enfermedades, incluyendo los géneros Borrelia, Rickettsia, Anaplasma, Ehrlichia, Francisella y Babesia. Asimismo, existen estudios que reportan una gran biodiversidad de comunidades microbianas presentes en diferentes especies de garrapatas (De la fuente et al., 2008; Almeida 2012; Carpi et al., 2011; Heise et al., 2010; Lalzar et al., 2012; Menchaca et al., 2013; Ponnusamy 2014). Por otro lado, diversos hallazgos muestran que las garrapatas transportan comunidades microbianas complejas, donde predominan los microorganismos simbiontes, relacionados con diversos procesos biológicos, de adquisición y transmisión de patógenos, fisiológicos y reproductivos. Estas investigaciones, muestran que muchas de las comunidades de bacterias no patógenas, pueden variar dependiendo de la especie de garrapata, la región geográfica de donde se colectan, la temporada, los estadios dentro del ciclo de vida, y su localización anatómica (Andreotti et al., 2011; Qiu et al., 2014; Swei and Kwan, 2017). Sin embargo, y a pesar de estos hallazgos, actualmente no se ha elucidado el papel y función que tienen estos microorganismos dentro del ciclo de vida de la garrapata, sumado a la gran limitante que representa, el aislamiento, cultivo y propagación en medios de cultivo de las bacterias que transmiten, como es el caso de Anaplasma spp. y Babesia spp.; además del conflicto que existe para la infestación con garrapatas de bovinos experimentales bajo condiciones controladas, lo que dificulta el aislamiento de la microbiota de cada tejido. A la fecha, y a pesar de estas limitantes, se han logrado identificar algunas especies de bacterias patógenas y su localización tisular, como es el caso de Anaplasma, Borrelia, Coxiella, Ehrlichia, Francisella y Rickettsia, especies consideradas de importancia médico-veterinaria, por su alto nivel de virulencia (Bonnet et al., 2017; Greay et al., 2018). Por otra parte, se ha identificado la presencia de bacterias comensales presentes en la piel del ganado bovino como es el caso de la bacteria Staphylococcus saprophyticus, cuya presencia también ha sido reportada en exudados de la garrapata R. microplus, actuando como bio-indicadores de enfermedad o infección, en donde la bacteria tiene un alto potencial patógeno para la garrapata hembra, ya que los individuos infectados, pierden la capacidad de ovipositar, provocando eventualmente su muerte (Miranda-Miranda et al., 2009 y 2010). Estos hallazgos, representan una aproximación en la búsqueda de nuevas estrategias de control natural, basadas en el uso de antagonistas de la propia garrapata que se encuentran presentes en la piel del hospedero bovino. Por otra parte, existen estudios que reportan la microbiota en diversas especies de garrapatas como Ixodes, Amblyomma, Dermacentor, Haemaphysalis y Rhipicephalus, revelando la presencia de diversas poblaciones de patógenos transmisibles como virus, bacterias y protozoarios (Narasimhan 2014). Evidencias recientes, han demostrado que muchas de las poblaciones microbianas pueden interactuar activamente de manera directa o indirecta con los patógenos que transmite la garrapata. Un ejemplo, se reportó en la especie Ixodes scapularis, donde se comprobó que la eliminación de la población bacteriana presente en el intestino medio, a través de tratamiento exacerbado de antibióticos en ratones utilizados como fuente de alimento, puede alterar de manera significativa la infección por Borrelia burgdorferi, agente causal de la Enfermedad de Lyme (Narasimhan 2014). Estas bacterias espiroquetas, disminuyen su capacidad infectiva en respuesta a dicho tratamiento. Por otro lado, las bacterias causantes de la anaplasmosis granulocítica humana, Anaplasma phagocytophilum, son capaces de reducir la viabilidad de poblaciones presentes en las garrapatas de ganado I. scapularis (Abraham et al., 2018). Esta bacteria, ingresa a la garrapata durante el proceso de ingestión sanguínea y una vez dentro, la bacteria migra rápidamente desde el intestino y se establece en las glándulas salivales, donde se mantiene durante las diferentes etapas del ciclo de vida del artrópodo (Noda et al., 1997; Yuval et al., 1990). A la fecha, no es claro cómo la bacteria penetra y coloniza el intestino medio de la garrapata, ni mucho menos, los mecanismos moleculares asociados. Sin embargo, se ha demostrado que la glicoproteína anticoagulante IAFGP de I. scapularis, es capaz de inhibir la formación de las biopelículas bacterianas (Heisig et al., 2014). Interesantemente, la bacteria A. phagocytophilum, secuestra a la proteína IAFGP, con el fin de alterar su acumulación en el intestino medio de las garrapatas para inhibir el desarrollo de biopelículas, permitiendo así su colonización y establecimiento en el vector (Heisig et al., 2014).
Los trabajos anteriores, remarcan la importancia del microbioma en el establecimiento de patógenos en el vector, así como su papel dentro de diversos procesos biológicos en el ciclo de vida de la garrapata. A pesar de la información generada, a la fecha no existe ningún reporte que demuestre el papel de la microbiota dentro de los procesos de capacidad vectorial, infección y trasmisión de patógenos (Fig. 1). Esto abre una extensa área de investigación para el desarrollo de trabajos novedosos con el fin de identificar y proponer blancos para el desarrollo de nuevos fármacos y/o vacunas contra las diversas enfermedades que afectan tanto al ser humano como a varios animales.
2. Factores abióticos y su influencia en el microbioma
La organización de la naturaleza en holobiontes, hace referencia a un tipo de organización que involucra a todas las especies, que de alguna u otra forma están interactuando y mantienen una estrecha comunicación. Sin duda, dentro de estas interacciones el medio ambiente juega un papel primordial en la regulación de la microbiota. Actualmente, se ha demostrado que la adaptación de los artrópodos a sus nichos, se encuentra fuertemente asociada a la composición de su microbioma, la cual, a su vez, puede responder diferencialmente a diversos factores abióticos (Minard et al., 2013 y 2015; Engel y Moran, 2013). El cambio climático global, ha obligado a la migración involuntaria de varias especies de garrapatas, lo que ha llevado a la colonización de nuevas áreas geográficas, que resulta en una disminución considerable en la diversidad de su microbiota (Minard et al., 2015). Por lo tanto, los factores abióticos juegan un papel determinante en la composición de su microbioma, ya que de éstos dependen las comunidades microbianas, las cuales, adquieren las funciones específicas necesarias para la adaptación y supervivencia del vector, así como de la adquisición y trasmisión de patógenos (Zouache et al., 2011; Minard et al., 2013; Villegas y Pimenta, 2014; Douglas, 2015). A la fecha, existen escasos reportes de la relación que existen entre los factores abióticos y la composición de la microbiota de vectores. Sin embargo, el mosquito Culicoides imicola, se ha reportado la presencia de endosimbiontes que forman parte de su microbiota, destacando que dicha composición y abundancia, se encuentra fuertemente vinculada a factores topoclimáticos donde llevan a cabo su reproducción (Campbell et al., 2004; Lewis et al., 2014). Adicionalmente, se han realizado caracterizaciones, donde se destaca el impacto de la humedad y la temperatura, en las poblaciones microbianas presentes en C. imicola, mostrando una baja diversidad de taxas en los mosquitos mantenidos en laboratorio bajo condiciones controladas, en comparación con los capturados en condiciones naturales (Douglas, 2011; Broderick y Lemaitre, 2012; Charan et al., 2016). Estos resultados demuestran el papel primordial que tiene el medio ambiente, evidenciando fuertemente la influencia de los factores abióticos sobre las comunidades microbianas que contienen los vectores artrópodos.
3. Aproximaciones ómicas en el estudio del microbioma de artrópodos
Actualmente, la secuenciación de siguiente generación y las tecnologías ómicas son herramientas valiosas para el estudio del microbioma. Los estudios metagenómicos y aunque en menor proporción los metatranscriptómicos, han sido utilizados para la caracterización de la microbiota de diferentes hospederos, y más recientemente de muchos vectores artrópodos de importancia médico-veterinaria (Greay et al., 2018). Así, la metaproteómica y la metabolómica, han surgido como poderosas herramientas que contribuyen a la elucidación de los diferentes procesos biológicos involucrados en las interacciones hospedero-vector-patógeno, y hospedero-vector-microbioma (Fig. 2) (Lou et al., 2017). En el caso particular de las garrapatas, los estudios integrativos de metagenómica, han contribuido a la caracterización de la microbiota de la garrapata Ixodes ricinus, que abarca más de 100 taxas, entre los que destacan, bacterias comensales, endosimbiontes y algunas patógenas (Hernández-Jarguín et al., 2018). El enfoque integrativo de estos análisis, sugieren la presencia de nuevos mecanismos en I. ricinus, por los cuales, la infección por patógenos afecta la formación de biopelículas que se encuentran relacionadas con la facilitación de la infección al hospedero (Hernández-Jarguín et al., 2018). Por su parte, el uso de los microarreglos o la tecnología de ARNseq, ha sido de gran ayuda para analizar los patrones de expresión del microbioma presente en garrapatas, y de evaluar su variabilidad bajo diferentes condiciones, con el fin de elucidar su papel dentro del vector (Fig. 2) (Rodríguez-Valle et al., 2018).
Figura 2.
Abordaje para el estudio del microbioma en la triada vector-microbioma-hospedero.
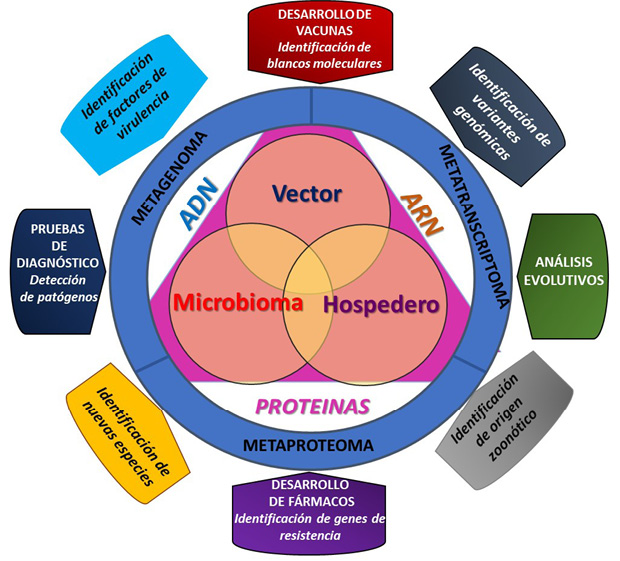
Las aproximaciones ómicas, basadas en ADN, ARN y proteínas, permiten el estudio de esta relación desde diversas perspectivas como son el desarrollo de fármacos, identificación de nuevas especies, desarrollo depruebas diagnósticas, etc.
4. Discusiones y conclusiones
Actualmente, las garrapatas se encuentran entre los vectores de transmisión de enfermedades más importantes, debido a la gran variedad de patógenos de importancia médica que transmiten a un amplio repertorio de hospederos animales, incluyendo al humano. Sin embargo, las garrapatas no sólo son portadoras de patógenos, sino también de un grupo de microorganismos comensales y simbióticos. En este sentido, la relación entre patógenos y el microbioma representa una caja negra de interacciones que a la fecha permanecen sin esclarecerse, y al mismo tiempo, es un área de investigación con un gran potencial para la identificación de posibles blancos para el desarrollo de vacunas contra diversas enfermedades. La microbiota de los artrópodos se relaciona con varios procesos biológicos como la adaptación, el desarrollo, la reproducción, la defensa contra el estrés ambiental y la inmunidad. Además del avance en los estudios del microbioma, existe información de cómo los patógenos son transmitidos y cómo pueden regular la presencia de las comunidades microbianas para facilitar su colonización en el vector. No obstante, aún es escasa la información científica acerca de las interacciones y mecanismos involucrados en la capacidad vectorial. Considerando que las interacciones biológicas funcionan como holobiontes (hospedador-microbioma), y de la hipótesis que la presencia de un patógeno puede alterar la composición microbiana dentro de una garrapata, resulta imposible pensar en la capacidad vectorial como un mecanismo aislado. En este sentido, los microorganismos simbiontes también pueden desempeñar un papel importante en la transmisión de patógenos.
Desde el punto de vista evolutivo, se sabe que muchos de los simbiontes poseen una historia conjunta con su hospedero de varios millones de años, lo que sugiere que estas asociaciones pueden haber evolucionado en interacciones más complejas que serían imposible de discernir sin analizar los componentes del holobionte. Estas aproximaciones genómicas basadas en filogenias, han ayudado a comprender mejor la historia evolutiva de los patógenos. Un ejemplo claro lo presentan miembros de Rickettsia, Francisella y Coxiella, que tradicionalmente eran reconocidos como patógenos vertebrados altamente virulentos, pero que ahora se proponen como microorganismos no patógenos que exhiben estilos de vida alternativos como mutualistas simbiontes dentro de la garrapata.
Desde hace varias décadas, varios investigadores han contribuido al conocimiento de las interacciones biológicas de los organismos. Basado en técnicas de bioquímica clásica, se ha podido identificar algunos microorganismos presentes en la cutícula de las garrapatas. Adicionalmente, a través de la secuenciación de Sanger, muchos científicos han identificado algunos genes que codifican para proteínas con potencial terapéutico, como la BM86 y algunas acuaporinas. Actualmente, las aproximaciones ómicas, basadas en ADN, ARN y proteínas, dentro del campo del control de plagas ha sido poco explorado, sin embargo, al momento contamos con diversas herramientas moleculares que nos han permitido elucidar mecanismos encaminados a la búsqueda de nuevos blancos. Dentro de la triada vector-microbioma-patógeno que conforma el holobionte, encontramos que el estudio de la metagenómica, la metatranscriptómica y la metaproteómica han sido herramientas fundamentales que han contribuido a la elucidación de mecanismos involucrados en esta interacción biológica. Estos estudios se centran en la identificación de factores de virulencia y variantes genéticos, en el diseño de pruebas diagnósticas, y en menor grado, en el desarrollo de vacunas y de nuevos fármacos para el control. No obstante, aún falta un gran trecho que recorrer, donde los estudios complementarios y en conjunto, parecen ser la clave para la erradicación de los patógenos de importancia veterinaria.
En conjunto, las garrapatas representan un sistema atractivo pero desafiante, en el cual, estudiar el microbioma y sus interacciones asociadas, representa un reto donde el conocimiento adquirido, permitirá la reevaluación de los riesgos para la salud, sin dejar de lado la importancia ecológica y evolutiva. Finalmente, el descifrar las relaciones entre los microorganismos presentes en las garrapatas, los patógenos que transmite y su hospedero, proporcionará información invaluable, que contribuirá al desarrollo de estrategias novedosas para el control de plagas de artrópodos y otros patógenos transmitidos por vectores.