Introducción
La influencia de la actividad humana en el antropoceno, es la causa principal del deterioro ambiental a nivel global. La agricultura intensiva, la industrialización, la rápida urbanización y el desarrollo de diversos productos como los xenobióticos, han generado un impacto notable en el ambiente (Lewis y Maslin, 2015). Los plaguicidas utilizados para el manejo de plagas en la agricultura, así como los antibióticos y los metales pesados son algunos de los xenobióticos que se liberan al ambiente, provocando efectos adversos sobre la salud humana y la biota de los ecosistemas. El autor Stephen Palumbi (2001), menciona que la mayor presión evolutiva sobre otras especies es ejercida por las actividades antropogénicas y considera que “los humanos somos la mayor presión evolutiva en del mundo”. Sin embargo, la variación genética y la microbiota de los individuos expuestos influye en cómo responde el huésped a la exposición de xenobióticos. En esta revisión abordamos el impacto de los diferentes xenobióticos en el ambiente y destacamos la importancia de la microbiota asociada a los organismos expuestos. Así como el potencial que tiene el hologenoma para contender a la exposición a xenobióticos, desde un contexto de superorganismo u holobionte.
1. Definiendo el hologenoma
En la naturaleza las relaciones simbióticas son ubicuas y su impacto ha sido trascendental en la evolución, diversificación de los organismos eucariotas y esencialmente en el éxito de la conquista de diversos ambientes, incluso adversos a los organismos (Angelard y Bever, 2013). La asociación con microorganismos puede ser benéfica, perjudicial o neutral y en algunos casos proveen al hospedero de nuevas capacidades metabólicas. De manera colectiva se define como “microbiota” a los microorganismos que viven en un ambiente definido, aunque en un principio el término se refería a los microorganismos que habitan exclusivamente el intestino humano. Por su parte, el término “microbioma” se refiere a todo el material genético de la microbiota (Lederberg y McCray, 2001; Marchesi y Ravel, 2005). Sin embargo, en años recientes ha tomado suma importancia el estudio de las interacciones entre los hospederos y sus socios microbianos, pero no como unidades autónomas sino como un súper organismo (compuesto por el hospedero más sus socios microbianos) llamado “holobionte”. Así la suma de la información genética del hospedero y más la de sus microorganismos simbiontes se denomina “hologenoma” (Zilber-Rosenberg and Rosenberg, 2008; Bordenstein y Theis, 2015; Simon et al., 2019).
2. Contaminación por xenobióticos
Los xenobióticos son sustancias químicas extrínsecas al metabolismo del organismo expuesto (Croom, 2012; Spanogiannopoulos et al., 2016). Este término es comúnmente utilizado para referirse a los químicos sintetizados por el hombre, que incluyen muchos químicos recalcitrantes, considerados un problema grave de salud pública y ambiental. También pueden incluir sustancias que se encuentran en la naturaleza, cuya concentración se incrementa por encima de lo usual, incluyendo los derrames industriales o derrames de petróleo (Kanapp y romley-Challoner 2003). Además del origen antrópico, y en un sentido menos estricto, también se consideran xenobióticos a aquellos sintetizados por algunos organismos como parte de su sistema de defensa, un ejemplo son las toxinas bacterianas, las micotoxinas y las toxinas producidas por plantas (Soucek, 2011; Abdelsalam et al., 2020). En la actualidad, la exposición a los xenobióticos es prácticamente ineludible y los efectos adversos son ampliamente reconocidos tanto en la salud humana y el ambiente. De manera cotidiana, se da el consumo de alimentos como frutas y vegetales, agua y bebidas como refrescos, vinos y jugos con residuos de xenobióticos. Sin embargo, la exposición a los xenobióticos también puede ser voluntaria, como el caso de los medicamentos y los antibióticos (Nicolopoulou-Stamati et al., 2016). En las últimas décadas, la contaminación por xenobióticos ha incrementado notablemente por la urbanización y las actividades humanas, principalmente en agua y suelo. Las fuentes de contaminación por xenobióticos son diversas e incluyen (I) la industria química y farmacéutica, (II) la industria del blanqueamiento de la pulpa del papel, (III) liberación de residuos de la industria minera como metales pesados, (IV) derrames accidentales de la industria petrolera y (V) la liberación de agroquímicos como plaguicidas y fertilizantes en la agricultura intensiva (Singh 2017; Embrandiri et al., 2017).
2.1. Plaguicidas
La agricultura convencional depende en gran medida del uso de fertilizantes y plaguicidas para maximizar la producción y por ende incrementar las ganancias económicas (Aktar y Chowdhury 2009). Después de la Segunda Guerra Mundial, la necesidad de incrementar el rendimiento de los cultivos para cubrir la demanda creciente de alimentos, propició el uso de estos agroquímicos (Popp et al, 2013; Javaid et al, 2016). La Agencia de Protección Ambiental de Estados Unidos (EPA, por sus siglas en inglés) define a los plaguicidas como sustancias o mezclas de sustancias utilizadas para destruir, repeler o mitigar cualquier plaga. Estos productos pueden ser sintetizados químicamente o de origen natural. Se utilizan comúnmente en la agricultura para proteger a los cultivos, principalmente de hierbas e insectos plaga y en el área de la salud pública para el manejo de vectores transmisores de enfermedades como la malaria, fiebre del dengue, chagas, entre otras (Rose, 2001; Blair et al., 2015). Particularmente en la agricultura, las pérdidas por daños causados por plagas son de aproximadamente 45%. Sin embargo, los plaguicidas son la manera más rentable para incrementar el rendimiento de los cultivos hasta en un 30% a nivel mundial (Bromilow, 2005). El uso de los plaguicidas incrementó notablemente a partir de los años 90s y se estima que 2 millones de toneladas de plaguicidas son utilizados anualmente en todo el mundo. Se estima que para el año 2020 el uso mundial de plaguicidas incremente a 3.5 millones de toneladas anuales (Sharma et al, 2019). Específicamente en Estados Unidos de América, se utilizan anualmente 500 millones de kg de plaguicidas, con un costo que asciende a los 10 billones de dólares por año (Pimentel et al., 1998; Sharma et al., 2019). En el mercado mundial, esto representa entre el 16 y 18% del total de plaguicidas, los grupos de plaguicidas mayormente utilizados por EE.UU. son herbicidas (53%), insecticidas (14%) y fungicidas (10%). Por su parte, México es el tercer país con mayor demanda de plaguicidas en América del Norte, después de EE. UU y Canadá. Se estima que para el periodo de 2017-2022, México incremente su demanda aproximadamente 5.2%. Los principales grupos de plaguicidas utilizados en México son herbicidas e insecticidas que comprende el 36% del mercado de América del Norte. Uno de los plaguicidas más utilizados entre los años 1997 y 2000 fue el DDT (dicloro difenil tricloroetano), un compuesto organoclorado usado como insecticida principalmente contra vectores de enfermedades como dengue, malaria, fiebre amarilla y tifus. Aunque desde 1962 Rachel Carson advertía sobre los efectos adversos de su uso en la Primavera Silenciosa, fue hasta el año 2000 que se prohibió el uso del DDT en México (Sharma et al., 2009; Abhilash y Singh, 2009). Sin embargo, en México y en muchos otros países el uso de otros plaguicidas peligrosos sigue en curso. Entre los principales riesgos del uso de plaguicidas se encuentran: 1). La exposición ocupacional, es decir cuando los trabajadores se encuentran expuestos en su línea de trabajo (agricultores, exterminadores de plagas, jardineros, trabajadores de la industria de los plaguicidas); 2). Exposición de la población (mediante el agua o alimentos contaminados con plaguicidas). Además de los riesgos sobre la salud humana, múltiples evidencias indican que los plaguicidas pueden ocasionar 3). Efectos adversos sobre el ambiente (contaminación del agua, suelo y aire, efectos sobre organismos no blanco: plantas, mamíferos, peces, insectos, etc.) (Damalas y Eleftherohorinos, 2011). Además, el uso repetido del mismo grupo de plaguicida, así como la aplicación de dosis excesivas actúa como una presión de selección sobre la población del organismo plaga, generando cambios en el pool genético e incrementando la población de los individuos resistentes (Kranthi et al., 2002; Naveen et al., 2017). La resistencia a los plaguicidas es definida por el Comité de Acción de Resistencia a Insecticidas (IRAC, por sus siglas en inglés) como un cambio heredable en la sensibilidad de la población de una plaga y se refleja como el fracaso repetido de un producto para lograr el nivel de control esperado cuando se utiliza de acuerdo con las recomendaciones para ese organismo plaga (https://irac-online.org/). Los mecanismos de resistencia a plaguicidas son variados, estos incluyen 1) Resistencia por modificación del sitio blanco: el sitio blanco en donde actúa el plaguicida es modificado y en consecuencia se reduce o elimina el efecto del plaguicida, 2) Resistencia metabólica: se refiere a la capacidad metabólica del organismo para la degradación o detoxificación del plaguicida de manera más rápida que los organismos susceptibles, la actividad catalítica de las enzimas involucradas en metabolizar o desintoxicar está muy relaciona a la resistencia. Entre ellas se encuentran las enzimas citocromo P450, que son un grupo de monooxigenasas que participan en el metabolismo de xenobióticos como: plaguicidas, medicamentos y toxinas vegetales. Estas monooxigenasas, se encuentran en los organismos aerobios como insectos, plantas, mamíferos, aves y también en bacterias. Las monooxigenasas oxidan una amplia variedad de compuestos. Por otro lado, las enzimas Glutation transferasas confieren la resistencia metabólica mediante el secuestro del plaguicida. Es importante mencionar que los simbiontes también son considerados un mecanismo de resistencia, principalmente relacionada a la resistencia metabólica, 3) Resistencia por modificación en la cutícula: la cutícula es una barrera frente a la exposición a los plaguicidas, ciertas modificaciones en la expresión de proteínas cuticulares reducen la penetración del plaguicida al insecto, este tipo de resistencia en conjunto con la resistencia metabólica, pueden conferir altos niveles de resistencia a plaguicidas, 4) Resistencia por transportadores ABC: los transportadores están involucrados en el transporte de moléculas como aminoácidos, azúcares, lípidos, péptidos con dependencia de la hidrólisis de ATP, se encuentran distribuidos en todos los organismos y se ha reportado que juegan un papel importante en la detoxificación de xenobióticos (Figura 1).
Figura 1
Mecanismos de resistencia a insecticidas
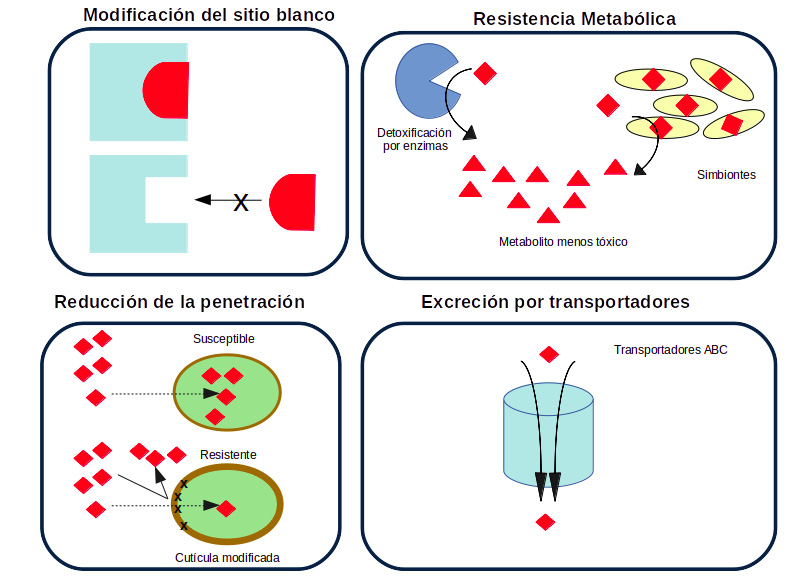
Modificado de Le Goff et al., 2019.
Sin embargo, una amplia diversidad de microorganismos del suelo tienen la capacidad de degradar xenobióticos, incluyendo a los plaguicidas. Estos organismos poseen características genéticas y enzimáticas que les permiten degradar a los plaguicidas en compuestos inorgánicos menos tóxicos (Verma et al., 2014). Las bacterias y los hongos son los principales degradadores. Entre los aislados bacterianos destacan Bacillus, Pseudomonas, Flavobacterium, Arthrobacter, Diaphorobacter, Klebsiella, Ochrobactrum, Agrobacterium, Burkholderia, Kocuria, Archromobacter, Sphingomonas, Chromohalobacter, entre otros que han sido reportados como degradadores de plaguicidas (Ataikiru et al., 2020; Inthama et al., 2021). Las condiciones ambientales, en particular la presencia o ausencia de oxígeno tiene una influencia notable en la degradación de plaguicidas (Reineke, 2001). Los ambientes aeróbicos se caracterizan por la presencia de oxígeno libre (O2), en estos ambientes los microorganismos aerobios crecen y llevan a cabo su metabolismo en presencia de oxígeno molecular, realizan la degradación a través de reacciones oxidativas. En contraste, los ambientes anaerobios carecen de oxígeno molecular y los microorganismos anaerobios realizan la degradación a través de reacciones reductoras (Reineke, 2001).
Los microorganismos pueden utilizar a los plaguicidas como una fuente de nutrientes. Primero el plaguicida entra al microorganismo y posteriormente a través de reacciones enzimáticas lo degradan en moléculas pequeñas no tóxicos o de menor toxicidad (Huang et al., 2018). La capacidad de los microorganismos para la degradación de plaguicidas y su capacidad para adaptarse a ambientes contaminados amplían las capacidades fisiológicas del hospedero y ofrece un gran potencial para contender ante la exposición a plaguicidas (Werren et al 2012; Blanton y Peterson, 2020)
2.2. Metales pesados y metaloides
Un grupo importante de xenobióticos son los metales pesados y los metaloides, ambos son elementos inorgánicos. Los metales poseen una gravedad específica cinco veces mayor que el agua, por su parte los metaloides son sustancias intermedias entre metales y no metales, son semiconductores y no presentan lustre, por ejemplo, el arsénico, boro y el silicio (Tchounwou et al., 2012). Ambos se encuentran naturalmente en bajas cantidades en el suelo, en el agua y en la atmósfera, incluso algunos tienen funciones biológicas importantes: cofactores de enzimas y participan en procesos de oxido-reducción. Sin embargo, las actividades antropogénicas contribuyen a incrementar las concentraciones de estos xenobióticos en el ambiente, ocasionando un riesgo para la salud humana y los ecosistemas (He et al., 2005). Los metales y metaloides son los principales xenobióticos que se acumulan en el suelo, derivado de diferentes actividades: son utilizados en el sector agrícola como plaguicidas o fertilizantes, también se utilizan en la industria maderera para protección de la madera, en la industria del vidrio, en la industria petrolera y de manera considerable la industria minera. Esta última genera grandes cantidades de residuos sólidos conocidos como “jales mineros”, resultado de la extracción de los minerales (Blowes et al., 2003; Chung et al., 2019). Anualmente se producen 25 mil millones de toneladas de residuos sólidos derivados de las actividades mineras, generando un impacto negativo en los ecosistemas y en la biodiversidad (Mudd y Boger, 2013; Murguía et al., 2016). Los metales pesados resultan ser tóxicos a altas concentraciones para la mayoría de los organismos, en las plantas pueden ocasionar efectos negativos que incluyen una baja en la acumulación de biomasa, clorosis, baja asimilación de nutrientes, desequilibrio hídrico, inhibición del crecimiento y fotosíntesis, senescencia y la muerte (Singh et al., 2016). Sin embargo, algunas han seleccionado mecanismos que les permiten colonizar ambientes con altas concentraciones de metales pesados. En las plantas, la “rizosfera” representa la interfaz entre la planta y la raíz, es el área alrededor de la raíz que se encuentra colonizada por una población específica de microorganismos, resultado de la atracción por los químicos o exudados liberados por la raíz de la planta, el término fue propuesto por el alemán Lorenz Hiltner (1904). Esta asociación con microorganismos puede permitirles contender contra diversos factores ambientales o situaciones de estrés. Por ejemplo, las plantas tolerantes a metales pesados ofrecen en la rizosfera condiciones atractivas para ciertas comunidades microbianas que contribuyen o confieren la tolerancia a los metales pesados, por acumulación, desintoxicación o transformación (Mishra et al., 2017). Además de la capacidad de las plantas para resistir a los metales pesados, los microorganismos como las bacterias endófitas, rizobacterias y hongos han tomado gran interés debido a que incrementan la tolerancia a metales pesados en plantas (Franco-Franklin et al., 2021). El potencial de las plantas tolerantes y microorganismos resistentes a metales pesados ha dado lugar al uso de tecnologías como la fitorremediación, utilizando a la planta y a sus bacterias simbiontes para descontaminar suelos con metales pesados (Rajkumar et al., 2012; Kong et al., 2017; Mesa-Marín et al., 2020). Los mecanismos para la captación de los metales pesados incluyen: 1) La bioactivación del metal en la rizosfera por interacciones entre microbios con la raíz de la planta, mediante la alteración de la permeabilidad de la membrana de la planta, cambiando la capacidad de unión de las membranas celulares o por quelación por exudados. 2) Captación del metal por medio de transportadores de metales en la membrana plasmática, incluyen transportadores tipo ATPasa, difusión facilitada de cationes y resistencia natural por la familia de proteínas de macrofagos. 3) Detoxificación de metales por apoplastos, a través de la unión de las paredes celulares y la quelación del metal en el citoplasma por diferentes ligandos como fitoquelatinas, metalotioneínas y proteínas de unión a metales. 4) Secuestración de metales dentro de vacuolas por transportadores localizados en tonoplastos, los metales se concentran en las partes aéreas de la planta en concentraciones superiores a las concentraciones del suelo (Kapagunta, 2017).
2.3. Antibióticos
Los antibióticos son otro grupo de xenobióticos, ampliamente utilizados a nivel global como medicamentos para contender contra las infecciones en humanos, animales domésticos y de granja. Los antibióticos son sustancias que inhiben el crecimiento de las bacterias. De manera que matan o evitan que las bacterias se reproduzcan. Los antibióticos son sintetizados naturalmente por diversos organismos o son sintetizados químicamente por el hombre. Además de su uso contra infecciones bacterianas, los antibióticos se utilizan incluso como promotores de crecimiento animal (Gothwal y Shashidhar, 2014). Sin embargo, el uso desmedido de antibióticos y sus descargas en el ambiente ha tenido consecuencias notorias, sobre todo incrementando las poblaciones de bacterias resistentes, impactando en la estructura y diversidad de la microbiota ambiental y generando un serio problema de salud pública (Martínez, 2009; Kraemer et al., 2019). Particularmente en el sector pecuario, el uso de antibióticos es una práctica común, aunado a la creciente demanda y a la producción a gran escala en las granjas, esto podría derivar en un incremento en el consumo de antibióticos hasta en un 67% para el año 2030 (Van Boeckel et al., 2015). Únicamente en Estados Unidos de América el 80% de los antibióticos en venta se consumen por el sector pecuario, con el objetivo de prevenir infecciones y mejorar las tasas de crecimiento de los animales para consumo humano. Los antibióticos no son metabolizados completamente en el organismo y son excretados del cuerpo y dispersados en el ambiente, principalmente a través del agua, causando diversos efectos adversos como la contaminación de los ecosistemas y la alteración de diferentes funciones ecológicas como; cambios en la transformación del nitrógeno, metanogénesis, reducción del sulfato, ciclo de nutrientes y alteración en la degradación de la materia orgánica (Roose-Amsaleg y Laverman, 2016; Grenni et al., 2018). La contaminación por antibióticos causa una disminución en la biodiversidad, altera la actividad enzimática y la producción de biomasa (Grenni et al., 2018). Además, el uso de antibióticos con fines no terapéuticos en animales, favorece la resistencia a antibióticos en bacterias patógenas de humanos (Martin et al., 2015). Otro problema importante, es la transmisión de bacterias resistentes por exposición ocupacional, pues hay evidencia de que los entornos antropogénicos como las granjas, influyen sobre el microbioma y resistoma (la colección de genes de resistencia a antibióticos) intestinal de los trabajadores, causando un enriquecimiento de taxas potencialmente patógenos, encontradas también en los ambientes cercanos a las granjas, sugiriendo un intercambio entre el entorno y el personal que labora en las granjas (Sun et al., 2020). Estudios indican que, en humanos, la microbiota intestinal está involucrada, entre otros procesos, en la maduración del sistema inmune, modulación de procesos nutricionales y metabólicos (Le Chatelier et al., 2013; Gensollen, et al., 2016; Zheng et al., 2020). La transferencia de la microbiota intestinal humana de gemelos discordantes a ratones axénicos resulta en la transmisión del fenotipo metabólico (obeso o delgado) del humano a los ratones. De manera interesante se observó que la convivencia de ratones obesos y delgados impedía el desarrollo de adiposidad y masa corporal en los ratones obesos, esto se correlacionó con la invasión de Bacteroidales de los ratones delgados a los obsesos y en cierta medida el rescate del fenotipo también se correlacionó con la dieta, indicando que las interacciones de la dieta y la microbiota influencian la biología del hospedero (Ridaura et al., 2013). Los antibióticos también pueden causar la disbiosis de la microbiota y la acumulación de genes de resistencia (Francino, 2015). La resistencia a antibióticos en bacterias ha incrementado de manera notoria y se estima que para el año 2050 las infecciones causadas por patógenos resistentes a antibióticos cobrarán alrededor de 10 millones de vidas al año (O’Neill, 2016). Las bacterias utilizan dos estrategias para poder contender contra los antibióticos: 1) La resistencia mutacional, esto se da a partir del surgimiento de mutaciones que afectan la actividad del fármaco en bacterias que provienen de una población susceptible, generando una modificación del fármaco, reducción de la absorción, activando mecanismos de eflujo para expulsar al fármaco y 2) Adquisición de genes de resistencia por transferencia horizontal, (Munita y Arias, 2016). Entre los ejemplos de mecanismos de modificación del fármaco podemos mencionar modificación de los grupos amino o hidroxilo de aminoglucósidos por enzimas como las adeniltransferasas, acetiltransferasas y fosfotransferasas.
Los antibióticos es probablemente el xenobiótico más estudiado, en cuanto se refiere al efecto de estos sobre las comunidades bacterianas, ya sea por el desequilibrio que ocasiona en los sistemas, sobre todo en la microbiota intestinal o como la resistencia que genera (Greer et al. 2016). Pero también se han descrito la producción de diferentes compuestos antimicrobianos producidos por la microbiota de algunos sistemas (Offret et al. 2016). Lo anterior hace que la relación antibiótico-holobionte, sea un excelente modelo para el estudio y análisis de la evolución, funcionamiento e interacciones del hologenoma.
3. Discusión
3. 1. Influencia de la microbiota en la resistencia a xenobióticos
En el caso de los plaguicidas, existen numerosos reportes sobre la contribución de la microbiota del hospedero en la resistencia de insectos a plaguicidas (Paul y Mandal, 2019). Por ejemplo, Riptortus pedestris mantiene una relación simbiótica con Burkholderia, un simbionte adquirido del ambiente, se aloja en las criptas del intestino del insecto. Este simbionte facilita la degradación del fenitrotion (organofosforado) y contribuye a la resistencia a este insecticida (Kikuchi et al., 2012; Itoh et al., 2018). Por otra parte, Plutella xylostella una plaga importante de crucíferas a nivel mundial, presenta poblaciones resistentes a insecticidas. Además, hay una correlación con un aumento de Firmicutes y menos Proteobacterias en el intestino de las poblaciones resistentes versus la población susceptible. Se ha observado que durante la exposición a los plaguicidas la diversidad de la microbiota intestinal se modifica, específicamente Enterococcus sp. potencia la resistencia a clorpirifos a través de la vitamina C y ácido acetilsalicílico mediante la modulación del sistema inmune (Xia et al., 2013; 2018). En Spodoptera frugiperda también se ha observado que la estructura de la microbiota cambia cuando se exponen a diferentes plaguicidas, la cepa resistente de S. frugiperda es un reservorio de bacterias degradadoras de plaguicidas (Almeida et al., 2017). Los casos mencionados anteriormente indican que la microbiota está relacionada con la resistencia a los insecticidas ya sea a través de la modificación de la estructura o por la actividad específica de un simbionte, así como la plasticidad del holobionte para contender contra situaciones de estrés.
Particularmente, las plantas leguminosas se asocian con rizobacterias fijadoras de nitrógeno que proveen varios beneficios a la planta: promoción del crecimiento, productividad, tolerancia al estrés biotico y abiotico, incluyendo la tolerancia a metales pesados. Se ha observado que la leguminosa Crotalaria pumila considerada una planta metalífera, es decir crece en zonas contaminadas con metales pesados, se asocia con Methylobacterium sp., quien es el miembro más dominante del microbioma y responsable de promover la germinación de las semillas, el desarrollo de las plántulas y además se transmite verticalmente como endófito de las semillas aún en condiciones de estrés por metales pesados (Sánchez-López, 2018). Las bacterias resistentes a metales pesados resultan una fuente potencial para el estudio de los genes involucrados en la resistencia. En el caso de S. meliloti CCNWSX00200 simbionte de Medicago lupulina, se han identificado mediante análisis transcriptómico y generación de mutantes a seis genes involucrados en la resistencia al Cu y Zn: una oxidoreductasa multicobre (CueO), una proteína de la membrana externa (Omp), sulfito oxidoreductasa (YedYZ), además de tres proteínas hipotéticas (una de ellas similar a CusA) (Lu et al., 2017). Los mecanismos para la resistencia de bacterias a metales pesados incluyen cuatro mecanismos particularmente involucrados en la resistencia al Zn, Cd y Pb: 1) Inmovilización de metales pesados en la pared celular bacteriana por exopolisacáridos (EPS), por ejemplo, en Sinorhizobium meliloti los (EPS) son responsables de la inmovilización de Cu2+(Hou et al., 2013). 2) Secuestro intracelular de metales pesados por proteínas quelantes, un ejemplo son las proteínas intracelulares SmtA tipo metalotioneínas, las cuales son proteínas ricas en residuos de cisteína que producen sideróforos responsables de la captación de los metales, 3) Secuestro extracelular de metales pesados por precipitación de formas insolubles a través de la excreción de sulfuros y fosfatos que precipitan al Cd y evitan su ingreso a las células, por ejemplo Klebsiella aerogenes y Citrobacter, respectivamente (Macaskie et al., 2000) y 4) Transporte activo de metales pesados por bombas de eflujo, que consisten de proteínas con un dominio transmembrana que transportan iones metálicos del interior como Cu+/+2, Ag+, Zn+2, al medio extracelular utilizando la hidrólisis del ATP (Lebrazi et al., 2018). En los casos mencionados la relación entre plantas y sus bacterias simbiontes va más allá de la fijación del nitrógeno y promoción de crecimiento, ya que pueden proteger al hospedero de los efectos por la exposición a metales pesados.
4. Conclusiones
Los organismos normalmente no aparecen aislados, sino relacionados entre sí con efectos sobre su eficacia biológica. Un holobionte es una forma de organización biológica que resulta de la asociación entre un organismo pluricelular, en el rol de hospedero, junto con su microbioma simbiótico. El microbioma se refiere al conjunto de microorganismos (fundamentalmente bacterias, pero también hongos, virus, etc.) que cohabitan simbióticamente junto con las células del organismo pluricelular (Carrier y Reitzel, 2017). La evolución en las técnicas de detección de microorganismos durante las dos últimas décadas ha revelado que los organismos pluricelulares conviven con un número elevadísimo de microorganismos simbióticos, en algunos casos (como el humano) tan numeroso como las propias células somáticas (Nolorbe-Payahua, et al. 2020), para tener un panorama más claro de esto último consideremos que el genoma humano contiene alrededor de 20.000 genes, pero su hologenoma contiene > 33 millones de genes aportados por su microbiota (Lloyd-Price et al., 2016).
Sin lugar a duda el desarrollo de las técnicas moleculares, la bioinformática y las tecnologías NGS ha jugado un papel relevante en la descripción de las comunidades microbianas como habitantes clave de los macroorganismos y como actores en los procesos biológicos, ecológicos y evolutivos; incluso si existen pocos estudios en cuestiones funcionales, en organismos modelo y no modelo y en todos los ecosistemas; ya sean terrestres, marinos o acuáticos, y que abarcan diferentes tipos de asociaciones huésped-microbiota ya sean laxas o estrechas (Bredon et al., 2018).
Se ha establecido por diferentes estudios el camino evolutivo del hologenoma y los holobientes, dónde van cambiando y se van ajustando de acuerdo al paso de la historia evolutiva. A medida que los hábitats cambian debido a las presiones globales y locales, la resiliencia de la población y los procesos de adaptación dependen no solo de sus reservas genéticas, sino también de sus comunidades de bacterias asociadas (Suárez, 2020). Estas relaciones son tan estrechas que incluso se ha desarrollado la teoría evolutiva de unidad de selección, que asegura que el hologenoma resultante de esta organización se ha formado durante millones de años (Morris, 2018), pero también debe mantenerse estable soportando incluso las presiones globales y locales de sus hábitats (Aires y Engelen, 2016).
El progreso reciente en la comprensión de la influencia del hologenoma en la evolución, la ecología y la salud de su huésped, está directamente relacionada con en el desarrollo de herramientas adecuadas para caracterizar y estudiar mejor el proceso de ensamblaje y el funcionamiento de las asociaciones huésped-microbiota. En estudios recientes utilizando la metatranscriptómica se caracterizó la microbiota asociada a las raíces de Holcus lanatus y sus cambios en relación con la composición del suelo. Específicamente, se detectaron cambios en las comunidades de hongos micorrízicos arbusculares de acuerdo con la disponibilidad de fósforo en el suelo (Young et al., 2018).
De forma similar, Cregger y colaboradores (2018) analizan el holobionte de Populus, caracterizan los efectos respectivos de los tejidos y genotipos vegetales sobre la estructura de la comunidad microbiana. Este estudio proporciona una comprensión holística de la estructura del microbioma dentro de una planta hospedante relevante para la bioenergía. Como tal, constituye un atlas o mapa detallado para más pruebas de hipótesis sobre la importancia de taxones microbianos individuales dentro de nichos y hábitats específicos de Populus y una línea de base para comparaciones con otras especies de plantas (Cregger et al., 2018). Lo anterior muestra que el análisis funcional basado en metatranscriptomas es una herramienta poderosa para el estudio de las interacciones naturales entre plantas y microbiomas, que sin lugar a dudas proporciona un potencial enorme en trabajos en control de plagas o en la tolerancia a diversos xenobióticos.
Los hologenomas resultados de estos procesos evolutivos, identificados en todos los sistemas que se han mencionado con anterioridad, adicional a su amplio poder de resiliencia, son potencialmente un reservorio casi infinito de genes que pueden funcionar como una sola maquinaria para contender con la presencia de xenobióticos resultantes de las actividades antropogénicas o en algunos casos, como los antibióticos, poder sustituirlos por compuestos que generan menos resistencia y un menor impacto a los sistemas. Como se mencionó, existen diversos ejemplos de mecanismos por los cuales los microorganismos asociados pueden degradar, eliminar, transformar o eludir a estos compuestos.
Otro punto importante, se relaciona con el proceso de ensamblaje de la microbiota y los mecanismos utilizados por el huésped para controlar el reservorio y el mantenimiento de sus socios microbianos. Al alterar experimentalmente la microbiota asociada con Daphnia mediante un tratamiento con antibióticos e inocular a individuos libres de gérmenes con la microbiota alterada, Callens y colaboradores (2018) muestran que la alteración inducida por antibióticos afecta fuertemente al ensamblaje de holobiontes con consecuencias sobre el crecimiento del huésped. Esta alteración provocada por antibióticos tuvo un fuerte efecto en la colonización posterior de Daphnia al afectar las interacciones ecológicas entre los miembros de la microbiota. Demostrando que la composición del grupo de microbiota colonizadora puede ser un factor estructurante importante del ensamblaje de la microbiota en Daphnia, afectando la composición de holobiontes y el crecimiento del hospedador. Estos hallazgos contribuyen a una mejor comprensión de cómo el entorno microbiano puede dar forma a la composición del holobionte y afectar las interacciones huésped-microbiota.
Por otro lado, Vannier y colaboradores (2016) analizan la transmisión de la microbiota asociada a la planta Glechoma hederacea durante la propagación vegetativa. Descubrieron que una proporción significativa de las comunidades microbianas de las plantas madre se transmitían a sus hijas a través de conexiones entre individuos. Lo que ejemplifica la transmisión vertical del hologenoma y su potencial que tiene en la biotecnología aplicada a los cultivos vegetales.
Brener-Raffali y sus colegas (2018) demostraron que tanto los factores genéticos como los ambientales dan forma a la composición microbiana y destacaron en particular la influencia de la temperatura del mar y sus consecuencias sobre la resiliencia de los corales en un contexto de calentamiento global. También informaron del descubrimiento de productos naturales con actividades reguladoras que pueden tener aplicaciones biomédicas potenciales, con lo que se reafirma el reservorio inherente en las comunidades microbianas para el desarrollo de nuevos fármacos.
En estudios similares, Ravanbakhsh y colaboradores (2018) demuestran el papel de la microbiota en la señalización del etileno de plantas, una vía importante en los mecanismos de defensa de las plantas. Guégan y colaboradores (2018) destacan la importancia de la microbiota de insectos en mosquitos, analizan el potencial del holobionte del mosquito para nuevas estrategias para el control de vectores.
Un interesante estudio proporciona nuevos conocimientos sobre la contribución de la microbiota a la digestión de fuentes de alimentos terrestres, lo que puede haber permitido la colonización de la tierra por isópodos terrestres. Se identificaron un repertorio de enzimas complementarias para la degradación de lignocelulosa tanto del huésped como del microbioma en A. vulgare. Lo que proporciona nuevos conocimientos sobre el papel del microbioma en la evolución de los isópodos terrestres y su radiación adaptativa en los hábitats terrestres (Bredon et al., 2018). Este tipo de estudios abre una gran ventana de posibilidades en la búsqueda de enzimas que puedan contribuir en la industria.
Aunque existen diversos ejemplos en los que utilizan estas maquinarias para resolver problemas ambientales, estos se reducen al uso de genes de manera individual o de algunas bacterias o en el mejor de los casos consorcios bacterianos o mezclas de enzimas, dejando el enorme potencial que representa el TODO. Por lo que resulta imprescindible para el aprovechamiento del hologenoma, el entendimiento de su poder adaptativo, de su funcionamiento y de la complejidad en sus interacciones, para que después puedan ser utilizados como un arsenal en las estrategias para aminorar o reducir el impacto de los xenobióticos.
Agradecimientos
Se agradece al CONACyT por su apoyo al realizar este trabajo.
Fuentes de financiamiento
SEP-CONACyT Proyecto: A1-S-22034. Beca otorgada a RSM No. 296934
Disponibilidad de datos
Los autores declaran que todos los datos utilizados en la investigación estarán disponibles y sin restricciones de acceso.