1. De organismos a holobiontes
Hace apenas una década, poco se sabía sobre el gran repertorio de especies de microorganismos que habitan en todas las plantas y animales, sus redes de comunicación en los diferentes niveles de complejidad y la función ecosistémica que aportan al hospedero. Los recientes avances tecnológicos en la colecta y análisis de las secuencias de material genético han permitido el abordaje de estos microorganismos por primera vez en la historia. Resultado de este desarrollo científico, hoy podemos decir que un organismo multicelular ya no puede ser concebido como una entidad autónoma, formada únicamente de células producto de su genoma, sino más bien como redes biomoleculares compuestas por el huésped más sus microbios asociados, los cuales habitan a manera de simbiosis mutualista; es decir, sabemos ahora que somos unidades funcionales llamadas holobiontes (Bordenstein & Teis, 2015; Zilber-Rosenberg & Rosenberg, 2008).
Partiendo de este concepto, cada ser humano es entonces un ecosistema compuesto de millones de individuos pertenecientes a miles de especies de microorganismos, llamados en conjunto microbiota, o microbioma cuando se habla de los genes que conforman todo este ecosistema (Rojo et al., 2017). La mayor parte de esta comunidad de microorganismos son bacterias que residen en el intestino; en conjunto se le llama microbiota intestinal (MI) y puede incluir más de 3000 especies. La abundancia y diversidad varía de persona a persona hasta en 80% (Rojo et al., 2017; Ursell et al., 2012).
Muchas bacterias son específicas de la especie humana, por lo que se plantea que a lo largo de la historia han coevolucionado el Homo sapiens y diferentes especies de microorgansimos que conforman la microbiota (Moeller et al., 2016; Davenport et al., 2017). Algunos géneros que forman parte de la MI, aunque no son específicos del Homo sapiens, son Bacteroides, Bifidobacterium, Enterococcus, Prevotella, Lactobacillus, Clostridium, Faecalibacterium, Blautia, entre otras (Davenport et al., 2017).
Los miles de años de algunas asociaciones han brindado amplias oportunidades para que nuestra biología y la de los microorganismos hayan coevolucionado (Sonnenburg & Sonnenburg, 2019a). Evidencia de esto son las conexiones íntimas recientemente reveladas entre la MI y diferentes sistemas.
La MI puede sintetizar más péptidos y proteínas que los que están codificados en el material genético humano, superando su capacidad de síntesis en 100:1; participa en la biosíntesis de algunas vitaminas, la fermentación de polisacáridos complejos, la absorción de iones, la síntesis de neurotransmisores y la regulación de rutas metabólicas (Rojo et al., 2017; Guarner & Malagelada, 2003). Además, la MI produce numerosos péptidos antimicrobianos que regulan el crecimiento de las poblaciones de otros miembros del ecosistema, así como de microorganismos patógenos externos, y no solo bacterianos, sino también de parásitos intestinales (Ramírez-Carrillo et al., 2020), o incluso virales como el Sars Cov-2 (Yeoh et al., 2021), el virus que ha puesto en jaque a las poblaciones humanas y los sistemas de salud pública en todo el mundo. Se encontró, por ejemplo, que pacientes infectados con este virus carecían de grupos bacterianos con importantes funciones inmunomoduladoras, como Faecalibacterim prausnitzii, Eubacterium rectale y distintas especies de Bifidobacterium spp; por esto se concluyó que la MI es fundamental para la regulación del virus y de su sintomatología (Yeoh et al., 2021).
Más importante aún, la MI también regula el desarrollo, función y adaptación del sistema inmunológico del hospedero (Negi et al., 2019) y tiene grandes implicaciones en la comunicación y funcionamiento del sistema nervioso central (Borre et al., 2014; Ramírez-Carrillo et al., 2020).
Ahora bien, la MI está configurada como un sistema dinámico, es decir, que es susceptible de modificarse a lo largo del tiempo y que es vulnerable a distintas presiones de selección impuestas por el hospedero a lo largo de su vida (Walter & Ley, 2011). Los primeros pasos que permiten una diversidad ecosistémica microbiana ocurren incluso antes del nacimiento, pero sobre todo al momento del parto, donde estos microorganismos compiten por colonizar las superficies corporales expuestas al medio ambiente. Este proceso de desarrollo y diversificación estará influenciado por muchos factores socio-ambientales; incluidos el modo de nacimiento, la nutrición, la exposición a patógenos y antibióticos y las interacciones sociales del hospedero con otros miembros dentro y fuera de su comunidad (Stewart et al., 2018; Bokulich et al., 2016).
2. Los cambios drásticos en la vida del Homo sapiens y sus efectos en la MI
Al inicio de su aparición como especie, hace aproximadamente 200-300 000 años (Richter et al., 2017), los seres humanos vivían en pequeños campamentos de pocos miembros, usualmente familiares, que obtenían sus recursos alimenticios exclusivamente de la caza y la recolección (Hublin et al., 2017). Durante los últimos 10 000 años, a partir del advenimiento de la agricultura, nuestra especie ha experimentado cambios de forma drástica en sus prácticas y estilos de vida, que han resultado en importantes presiones ecológicas para la MI (Snir et al., 2015), que nos ha acompañado durante la mayor parte de nuestra historia evolutiva (Modi et al., 2014; Sonnenburg & Sonnenburg 2019b).
Uno de los principales factores reguladores de la diversidad y composición de la MI es la dieta (David et al., 2014; Moya & Ferrer, 2016; Shepperd et al., 2018). Ésta se ha modificado sustancialmente con la producción masiva de alimentos, incrementando la disponibilidad de proteína y grasa animal. La industrialización alimentaria incrementó el consumo de sales y azúcares refinados, así como la incorporación de productos químicos coadyuvantes en los alimentos, como lo son los aditivos alimentarios entre los que se encuentran los conservadores y emulsificantes. A estos productos se suma el uso de pesticidas en la producción alimentaria. Todos ellos ejercen un profundo impacto en la dinámica ecosistémica de la MI (Monteiro et al., 2013; Sonnenburg & Sonnenburg, 2019b). Al mismo tiempo, estas dietas han disminuido la cantidad de carbohidratos complejos localizados en las fibras de plantas comestibles, como leguminosas, granos complejos, vegetales o nueces. Estos carbohidratos, conocidos como carbohidratos accesibles para la microbiota, MAC por sus siglas en inglés, son el principal recurso alimenticio y energético para nuestra MI (Sonnenburg & Sonnenburg, 2014). En modelos murinos se ha observado que una disminución de su consumo resulta en una extinción de especies microbianas en el curso de varias generaciones. Esto ilustra la importancia de los MAC en el mantenimiento del MI (Sonnenburg et al., 2016).
El desarrollo de antibióticos fue uno de los grandes avances que revolucionaron la medicina a finales del siglo XVIII y permitieron disminuir drásticamente las muertes ocasionadas por bacterias patógenas. Sin embargo, su uso también afecta a la MI. Se ha descrito que una sola profilaxis antibiótica puede diezmar y remodelar la MI, sobre todo con el uso de antibióticos B-lactámicos, como penicilinas y cefalosporinas (Zhernakova et al., 2016).
A la luz de este nuevo conocimiento, es posible que la guerra en contra de los microbios patógenos necesite reconsiderarse en términos que sean menos combativos. El gran éxito en el desarrollo de fármacos que eliminan de manera efectiva microorganismos patógenos y con mínimos efectos secundarios a corto plazo, ha llevado a su uso excesivo durante los últimos 30 años, lo que pudo haber afectado profundamente las dinámicas en el ecosistema de la MI. Aunado a esto, otras prácticas asociadas a la modernización en nuestro estilo de vida, como el incremento del parto tipo cesárea, la disminución de la alimentación por leche materna, las políticas de desinfección masiva, purificación de agua, drenaje, etc., son también factores que pudieron promover cambios de la MI (Zhernakova et al., 2016).
3. Microbioma ancestral y moderno
Si nuestro estilo de vida ha cambiado drásticamente, ¿debemos asumir que nuestra MI también? ¿En qué dirección? Responder estas preguntas es un gran reto metodológico, pues para hacerlo debemos involucrar el conocimiento histórico sobre la MI de sociedades pre-agrícolas o pre-industriales, lo cual no resulta sencillo de obtener. Una alternativa es mediante el estudio de coprolitos ancestrales localizados en asentamientos humanos que datan de estos periodos. Por ejemplo, uno de los pocos estudios que existen sobre coprolitos datados entre 3000 y 8000 años de antigüedad mostró que la composición de la MI durante estos períodos es más similar a las poblaciones con estilos de vida llamados “no-occidentales” o “no industrializados”, que a las poblaciones de las grandes ciudades industrializadas occidentales (Tito et al., 2012; Tito et al., 2008). Las poblaciones “no-occidentales” han sido definidas como pequeñas comunidades con prácticas sociales más cercanas a los estilos de vida de poblaciones ancestrales pre-industriales. Las características de este tipo de poblaciones incluyen obtención de alimentos mediante agroforestería a baja escala o caza-recolección, ausencia o bajo uso de antibióticos alopáticos, ausencia de mecanismos de sanitización y purificación de agua para consumo, bajo consumo de alimentos ultraprocesados, alimentación por leche materna durante los primeros años de vida, parto de tipo natural, etcétera (Barone et al., 2019). Aunque estas poblaciones tradicionales son de hecho genéticamente tan modernas como las industrializadas, es posible que la ausencia de las fuerzas selectivas para la MI, asociada a la urbanización, haya permitido que estas poblaciones alberguen una MI mucho más similar a la ancestral. Este enfoque de inferir características ancestrales a partir de poblaciones tradicionales toma mayor soporte al comparar poblaciones tradicionales alrededor del mundo que han estado aisladas durante decenas de miles de años (Sonnenburg & Sonnenburg, 2019a).
En la actualidad, poblaciones tradicionales con sistemas similares a los pre-agrícolas como los cazadores/recolectores del Pueblo yamomami del Amazonas (Clemente et al., 2015) o los Hadza en Tanzania (Smits et al., 2017) se caracterizan por una mayor diversidad en su MI que las poblaciones urbanas. Además, entre estas sociedades humanas se comparten algunos grupos taxonómicos que no existen en las sociedades industriales. Estos grupos microbianos se llaman volátiles o negativamente asociados con las sociedades humanas industrializadas, VANISH por sus siglas en inglés, se han encontrado de manera abundante tanto en muestras de coprolitos como en estas poblaciones tradicionales. Entre ellos se incluyen miembros de las familias Prevotellaceae, Succinivibrionaceae y Spirochataceae (Sonnenburg & Sonnenburg, 2019b).
Así como se muestra una disminución de estos grupos VANISH, las poblaciones industriales se han caracterizado por el aumento de otros grupos que generalmente no están o sólo se encuentran en bajas abundancias en poblaciones tradicionales. A estos grupos se les conoce como florecientes o seleccionados en sociedades de urbanización o modernización, BloSSUM por sus siglas en inglés, e incluyen a miembros de las familias Bacteroidaceae y Verrucomicrobia (Sonnenburg & Sonnenburg, 2019b), asociados a las dietas con alto contenido de azúcares refinados o a la exposición de antibióticos (Kindaichi et al., 2016). Estas diferencias también se han observado a nivel genómico, ya que muchos taxa VANISH presentan un gran repertorio de enzimas activas digestivas de carbohidratos (CAZymes) que se encargan de digerir polisacáridos complejos presentes en las fibras dietéticas. Por ende, parece ser que la escasez de MAC entre otras prácticas sociales en poblaciones urbanas disminuye la diversidad de la MI, extingue ciertos grupos taxonómicos, y promueve el aumento de otros (Sonnenburg et al., 2016).
En México, a pesar de contar con más de 56 grupos étnicos precolombinos con estilos de vida similares a los considerados tradicionales de otras partes del mundo, es muy poco lo que se ha estudiado sobre su MI en comparación con la población con estilos de vida occidentalizados. El único estudio realizado comparó un grupo indígena Me´phaa de la Montaña alta de Guerrero con una población urbana de la Ciudad de México. Aquí se encontró que la composición y abundancia depende de la población a la que se pertenece, así como del sexo y su grupo etario (Sánchez-Quinto et al., 2020). En general, la diversidad total es mayor en las poblaciones indígenas estudiadas que en la ciudad, además, presentan grupos exclusivos, lo que es especialmente evidente entre la cohorte de niños (Fig. 1; Sánchez-Quinto et al., 2020). Además, los niños de la ciudad presentan una fuerte dominancia de algunos grupos bacterianos asociados positivamente al metabolismo de azúcares refinados y a la exposición constante de antibióticos, como es el caso Saccharibacteria o miembros del filo Verrucomicrobia (Fig.1; Sánchez-Quinto et al., 2020). También se encontraron importantes diferencias en cuanto a los taxa VANISH, siendo el género Prevotella el de mayor abundancia en estas comunidades indígenas (Fig. 2).
Figura 1
Análisis Log2 fold-change de niños indígenas Me´phaa y niños de la Ciudad de México.
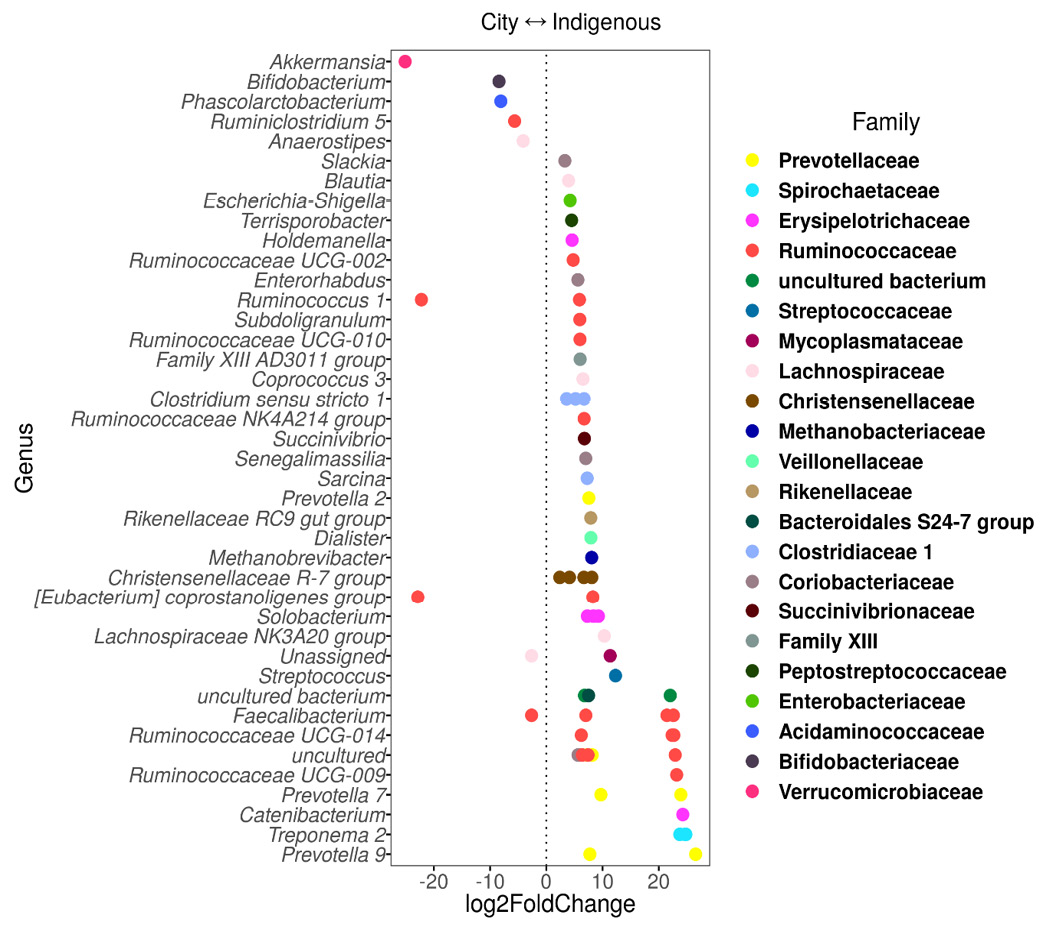
Fecal-prokaryotic ASVs agrupados por género y coloreado por familia (etiquetas). Los valores de Log2 fold-change indican la fuerza y dirección de la asociación hacia la ciudad (<0) y hacia los grupos Me’phaa (>0). Los géneros observados en esta figura fueron estadísticamente diferentes entre poblaciones a p < 0.01 corregido por el método FDR (false discovery rate). Reproducción de imagen autorizada explícitamente por los autores (Sanchez-Quinto et al., 2020).
Figura 2
Abundancia Relativa de los Grupos Bacterianos VANISH y BloSSUM entre niños de la Ciudad de México y Niños Indígenas (Me´phaa).
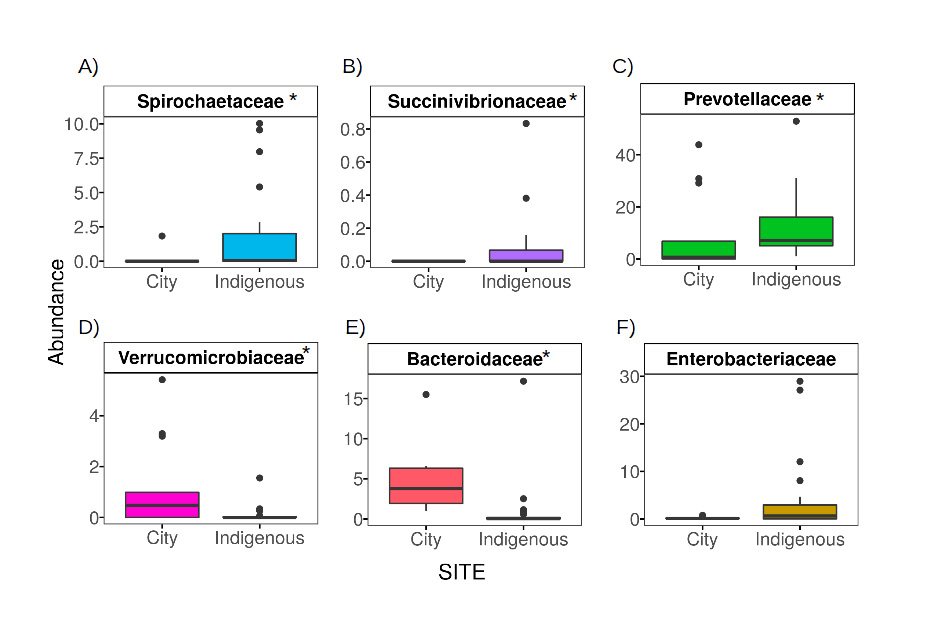
Los grupos VANISH se conformaron por las familias Spirochaetaceae, Succinivibrionaceae y Prevotellaceae (A–C); mientras que los grupos BIoSSUM por las familias Verrucomicrobiaceae, Bacteroidaceae y Enterobacteriaceae (D–F).
* Corresponde a diferencias significativas entre poblaciones a nivel de p < 0.01. Reproducción de imagen autorizada explícitamente por los autores (Sanchez-Quinto et al., 2020)
Por otro lado, las diferencias observadas entre sexos y edades en las comunidades indígenas se presentan en los grupos fuertemente vinculados al metabolismo de los MAC que se obtienen de dietas similares, por lo que se podría hablar de una redundancia funcional (Moya & Ferrer, 2016) presente en los indígenas, y que se ha perdido en la Ciudad de México (González-Santoyo et al., en preparación), es decir que los grupos indígenas presentan diferentes configuraciones de la MI pero que aportan las mismas funciones al hospedero. Los resultados de este estudio presentan importantes similitudes con lo observado en otras poblaciones tradicionales en el mundo y hacen suponer nuevamente este proceso coevolutivo entre la MI y Homo sapiens (Sánchez-Quinto et al., 2020).
4. Cambios en el MI y sus implicaciones en salud
¿Cuáles son las consecuencias de perder microorganismos que han sido nuestros simbiontes a lo largo de la mayor parte de nuestra historia evolutiva y que probablemente dieron forma a nuestra biología? ¿Podrían los microorganismos adquiridos recientemente, o aquellos con una mayor representación, conferirnos funciones redundantes o mejoradas en el contexto de nuestro entorno cambiante, en lugar de aquellos taxones ancestrales faltantes?, o ¿acaso este fenómeno generará nuevas interacciones que resulten en desafíos para la salud humana? Aún falta mucho por explorar para poder contestar de forma contundente cualquiera de estas preguntas. No obstante, cada vez se hace más necesario considerar a nuestra microbiota y sus genomas asociados, como un eje fundamental para la salud humana.
A la luz de este nuevo conocimiento, es claro que todos los cambios comportamentales en los estilos de vida moderna han limitado sustancialmente la exposición a microorganismos patógenos, han permitido el soporte alimenticio de una creciente población mundial y, en general, han promovido una mayor esperanza de vida. Sin embargo, y aunque resulte paradójico, estos beneficios pueden verse opacados por los costos en la calidad de vida en términos de salud. Como se ha mencionado antes, es necesario reconsiderar la guerra en contra de los microbios patógenos, ya que la aparición de superbacterias que son resistentes a los antibióticos y bactericidas químicos revelan ahora que hay un costo importante en nuestras estrategias para luchar contra estos microbios (Roca et al., 2015). Además, a largo plazo, y menos obvio, las alteraciones continuas en la MI debido a estas prácticas puede resultar en otros padecimientos crónicos no transmisibles (ENT, enfermedades crónicas no transmisibles) en la población humana, especialmente en edades adultas, el grupo poblacional que se vuelve mayoritario debido al incremento de la expectativa de vida (Sonnenburg y Sonnenburg, 2019a). Por ejemplo, a la fecha se han identificado al menos cincuenta ENT asociadas a cambios en la abundancia, composición y dinámicas de comunicación de la MI (Moya & Ferrer, 2016).
Estas ENT son consideradas una emergencia global, ya que son las responsables de 71% de las muertes que se producen en el mundo y el principal reto para la sostenibilidad de los sistemas de salud, de acuerdo con la “Agenda global 2030 para el desarrollo sostenible” de la OMS (Novelli et al., 2020). Por ejemplo, se estima que para 2030 la implementación de políticas públicas para prevenirlas y combatirlas podría ascender a más de 350 mil millones de dólares tan sólo en aquellos países con mayor incidencia (Bertram et al., 2019). Las ENT con mayor tasa de mortalidad y morbilidad son las enfermedades cardiovasculares, como los infartos al miocardio o la arteriosclerosis (Aronow et al., 2019), la diabetes (Pinilla et al., 2019), la mayoría de los cánceres como estómago, colon o sangre (Okamoto y Leischow, 2019), desórdenes neurodegenerativos, como esclerosis múltiple o Alzheimer (Rooney et al., 2017), y enfermedades respiratorias crónicas como asma o EPOC (Aggarwal, 2017).
En México, las muertes anuales por estos cuatro grupos representan el 60% de las muertes totales, siendo las enfermedades cardiovasculares (22%) y diabetes (16%) las de mayor incidencia (WHO, 2018). Estas cifras son de mayor relevancia si se considera que bastaron 20 años para colocarlas en la primera causa de muerte en nuestro país (WHO, 2018) y, según predicciones de la Organización Mundial de la Salud, continuarán en importante aumento si no se logra consolidar el conocimiento puntual de su etiología (WHO, 2018; Duan et al., 2018). Aunado a esto, la interacción con otros padecimientos transmisibles como la infección por el virus Sars Cov-2, ha resultado en un aumento dramático en las muertes de la población mundial durante 2020, ya que el primer factor que predice la intensidad de los síntomas, las complicaciones y las muertes por este virus, es precisamente las comorbilidades de las ENT más abundantes mencionadas arriba (Sanyaolu et al., 2020).
En un principio, se consideró que el aumento de las ENT era un resultado indirecto del franco incremento en la esperanza de vida de las poblaciones mundiales. No obstante, es difícil sustentar esta idea debido a que la mayor tasa de mortalidad por estas enfermedades corresponde a personas entre 30 y 69 años, siendo la primera causa de las denominadas muertes prematuras en personas adultas (WHO, 2018). En consecuencia, durante los últimos 15 años se han realizado grandes esfuerzos globales para conocer los factores de riesgo asociados a este conjunto de padecimientos. Como resultado, se propuso que las ENT pueden ser producto de una compleja interacción entre características de predisposición genética, como el componente detonador, y factores de riesgo tanto comportamentales (p. ej. tipo de alimentación) como ambientales (p. ej. contaminación; Novelli et al., 2020). No obstante, esta propuesta ha tenido al menos dos retos importantes que limitan de forma considerable su valor explicativo.
Uno de ellos arguye al poco conocimiento de los mecanismos que vinculan estas complejas interacciones en la mayoría de las ENT, y el otro hace alusión al poco efecto que se ha observado respecto a la predisposición genética en la aparición de estos padecimientos. Por ejemplo, la búsqueda de polimorfismos de un solo nucleótido (SNP, por sus siglas en inglés) en estudios de genoma completo (GWA, por sus siglas en inglés) sobre la obesidad, uno de síndromes metabólicos de las ENT más estudiados, ha explicado únicamente entre el 0.1 y 3% de la varianza fenotípica de este padecimiento, inclusive en distintas poblaciones humanas (Müller et al., 2018).
Estas limitaciones abren una importante puerta para el planteamiento teórico y práctico de estrategias alternativas interdisciplinarias que incluyan, por ejemplo, la ecología evolutiva de la especie y su microbiota asociado. Aunque resulta difícil cuantificar el impacto que ha experimentado el genoma humano debido a los cambios en los modelos alimenticios, la evidencia reciente sugiere que las variaciones genómicas han sido mínimas. Un ejemplo es la tolerancia a la lactosa (Lewens, 2017), y que éstas dependen principalmente de la distribución geográfica de varios grupos ancestrales pre-agrícolas particulares; por ejemplo, aquellos distribuidos en Eurasia, América y África (Yang & Fu, 2018). Considerando esta aparente disimetría entre la tasa de cambios genómicos y los acelerados retos socio-ambientales que ha enfrentado nuestra especie, surge entonces la posibilidad de que la microbiota, y particularmente la MI, juegue un papel fundamental.
La MI tiene la capacidad de producir y metabolizar componentes fundamentales para el cuerpo humano; como es el caso de los ácidos grasos de cadena corta (AGCC), butirato, propinato y acetato, todos ellos producto de la fermentación, y mediadores en la comunicación con otros sistemas, como el sistema nervioso mediante producción de neurotransmisores, o la activación de lipoproteínas que promueven la acumulación de lípidos en los adipocitos. Por lo tanto, la MI juega un papel importante en el desarrollo y modulación del sistema inmune del hospedero, un componente fundamental que se ve alterado en el desarrollo de las ENT (Sommer y Bäckhed, 2013). Por ende, es claro que la influencia de la MI trasciende los confines del intestino forjando conexiones íntimas con todos los aspectos de la biología humana, y convirtiéndolo en una fuerte candidata que podría haber mediado las respuestas del hospedero a los cambios experimentados durante su historia evolutiva, y subsecuentemente, sería un blanco relevante para las políticas de salud pública (Sonnenburg y Sonnenburg, 2019b).
Aunque ha sido controversial la caracterización de una MI que sea considerada sana, en la mayoría de las definiciones se describe que una amplia diversidad es un marcador preponderante (Rojo et al., 2017) y, como se mencionó anteriormente, este componente está vinculado positivamente a los modos más tradicionales de subsistencia, donde el consumo de MAC es mayor y la exposición a antibióticos es menor (Rojo et al., 2017).
En este sentido, las fuerzas selectivas que hemos impuesto sobre la MI podrían ser particularmente importantes para la salud humana. Por ejemplo, la carencia de la actividad enzimática de las CAZymes, presentes en grupos VANISH, se encuentra asociada a la aparición de ENT o diferentes cánceres (colon, estómago o intestino); así como una disminución de AGCC, como butirato, propinato y acetato, todos ellos producto de la fermentación de estos polisacáridos gracias a la actividad de las CAZymes (Byndloss y Baumler, 2018).
En adición, la aparición de otros grupos bacterianos que han sido favorecidos por estas nuevas prácticas de vida no necesariamente es benéfica para el hospedero. Por ejemplo, estilos de vida industrializados presentan una expansión de bacterias anaerobias facultativas del filo Proteobacteria, microorganismos asociados a numerosas ENT (Byndloss y Baumler, 2018).
Otros, como Saccharibacteria que fue encontrada en mayor abundancia en pobladores de la Ciudad de México y está asociada a dietas con alto contenido de azúcares refinadas, o miembros del filo Verrucomicrobia, como Akkermansia sp. (Sanchez-Quinto et al., 2020), un género que se alimenta primordialmente del lumen intestinal, la primera barrera física en contra de baterías patógenas y otros agentes dañinos (Gareau et al., 2008).
Dicho lo anterior, es notable que diferentes aspectos de nuestra identidad microbiana ancestral se pudieron haber extinto en poblaciones urbanizadas mientras que otros han aumentado su presencia de forma drástica. Estos cambios han resultado en una discordancia entre nuestro microbioma recientemente adaptado, y la adaptación mucho más lenta de nuestro genoma (Sonnenburg y Sonnengurg, 2019a). Los grupos extintos podrían haber sido parte fundamental de la biología humana, moldeando diferentes características de su genoma, tales como vías de señalización, codificación de receptores o elementos regulatorios a lo largo de su evolución. La pérdida de estas señales esperadas por el genoma humano podría resultar en una desregulación de importantes sistemas, incluyendo el inmune, del metabolismo o del funcionamiento del sistema nervioso, contribuyendo así al aumento drástico de las ENT que actualmente observamos (Blaser, 2017).
5. ¿Qué podemos aprender?
Por norma evolutiva, los animales, incluyendo el ser humano, rara vez están completamente adaptados a su medio, ya que el cambio ecológico o social al que nos enfrentamos día a día es inminente. Ya sea por la aparición de nuevos patógenos, como el virus SARS-Cov2, o cambios en la fuente de alimentación, el genoma humano debe adaptarse constantemente. A medida que surgen nuevas presiones selectivas, el ajuste de los genomas de una población humana estará siempre por detrás de estas presiones, ya que se requieren de varias generaciones para que ocurran los procesos evolutivos darwinianos.
Durante la mayor parte de la historia evolutiva de la humanidad, hemos vivido en un entorno en el que los alimentos ricos en fibra eran básicos en la dieta y los alimentos industrializados y ricos en calorías no estaban disponibles. Sin embargo, en las últimas décadas la MI se ha adaptado a novedosas presiones selectivas impuestas por la industrialización. Pero aún falta mucho por dilucidar en esta fotografía histórica entre la evolución de la MI y la evolución de nuestra especie, sobre todo en conocer si esta nueva configuración de la MI puede ser negativa para el genoma humano donde, a su vez, han coevolucionado los genomas de miles de microorganismos. Por ejemplo, los alelos humanos que promueven la retención de calorías, aunque son benéficos en un entorno donde las fuentes de alimentos eran menos confiables, podrían tener efectos negativos en nuestro entorno moderno rico en alimentos calóricos (McGarvey, 1994). De manera similar, los alelos asociados a la pro-inflamación que proporcionaron un beneficio para la salud contra los patógenos en nuestro pasado antiguo, pueden ahora estar mal regulados y promover enfermedades inflamatorias crónicas (Birnkworth y Barreiro, 2014). Si los efectos influyen en la supervivencia y reproducción de los hospederos, estos alelos también se enfrentarían a una presión selectiva para eliminarlos de la población. La menor frecuencia de alelos que permiten la retención de calorías en las poblaciones que luchan contra la obesidad proporciona evidencia de que el inicio temprano del síndrome metabólico y otras ENT pueden impulsar ajustes en el genoma humano de toda la población para adaptarse a un entorno alterado (McGarvey, 1994). Con el conocimiento de que las comunidades microbianas intestinales pueden influir en gran medida en diversos aspectos de la fisiología del huésped, incluidos su estado inflamatorio, se augura que los cambios repentinos y drásticos en la MI causarán enfermedades crónicas diversas y generalizadas y, en última instancia, cambiarán los alelos humanos que confieren supervivencia y reproducción. Con esto, concluimos que es necesario más trabajo, y tal vez tiempo, para determinar hasta qué punto nuestro camino evolutivo se acopla para adaptarse a nuestro estado fisiológico recientemente derivado y dictado por los microbios.
Agradecimientos
El autor HAD agradece al fondo INIFAP-Fondos Fiscales No. de Proyecto SIGI 13512934133 y al Centro Nacional de Investigaciones Disciplinarias en Salud Animal, INIFAP.