Introducción
La microbiota humana, está conformada por una amplia variedad de taxones microbianos eucariontes, procariontes y virales que colonizan nuestro cuerpo de manera simbiótica, desempeñado roles muy diversos e importantes para el mantenimiento de la homeostasis (Huttenhower et al., 2012), que van desde el metabolismo, hasta el desarrollo y activación del sistema inmunológico (Wang et al., 2017)
Si bien fue en la década de 1670 cuando Leeuwenhoek, haciendo uso de sus microscopios rudimentarios describió por primera vez la asociación entre los microbios y el ser humano, han pasado más de 350 años, marcados por grandes hitos como el cultivo microbiano y el análisis genético, que nos ha permitido mejorar nuestra comprensión acerca de la distribución y conformación de estos microorganismos en los diferentes nichos corporales y las funciones que desempeñan en el proceso salud-enfermedad (Methé et al., 2012).
Herramientas analíticas como la metagenómica [análisis de genes y vías metabólicas] (Gilbert & Dupont, 2011), la pirosecuenciación de genes ARNr [en especial de ARNr 16S] (Jumpstart Consortium Human Microbiome Project Data Generation Working Group, 2012) y la secuenciación de escopeta [múltiples secuenciamientos de fragmentos pequeños] (Methé et al., 2012; Ursell et al., 2012), nos ha permitido descubrir que los microorganismos que habitan en nuestros cuerpos interactúan activamente en la prevención, desarrollo y pronóstico de diversas enfermedades, entre ellas el cáncer.
1. Colonización microbiana
Formada por entre 10 y 100 billones de microorganismos, nuestra microbiota iguala casi en proporción al número de células en nuestro cuerpo (Sender et al., 2016), siendo los filos bacterianos los más abundantes (99,7%), seguido de los microorganismos eucariotas (0.3%), Arqueas (0.01%), y en mínimas proporciones los virus (Methé et al., 2012); todos ellos comienzan un proceso de colonización en distintos tejidos ya desde los primeros momentos del nacimiento, con la ruptura de las membranas amnióticas y el paso del neonato por el canal de parto (Bokulich et al., 2016; Galazzo et al., 2020).
Las conformaciones que tengan estas primeras colonias microbianas, serán trascendentales en las primeras etapas de vida del neonato, ya que de esta dependerán procesos fisiológicos como la maduración del sistema inmunológico (Bennek et al., 2019), la digestión y metabolismo de alimentos (Turnbaugh et al., 2006) e incluso el desarrollo cognitivo (Carlson et al., 2018). Situaciones atípicas como el parto por cesárea, la administración de antibióticos y la alimentación con sustitutos de la leche materna variarán la conformación de esta microbiota (Korpela et al., 2018), relacionándose a posteriori con la aparición de posibles trastornos metabólicos (Cani, 2019), enfermedades intestinales (Buttó et al., 2015), trastornos alérgicos (Stefka et al., 2014) y enfermedades autoinmunes (Cahenzli et al., 2013)
En los siguientes tres años vida, la composición y diversidad de la microbiota experimentará grandes cambios, para finalmente alcanzar una conformación que conservará durante toda la vida [eubiosis] (Koenig et al., 2011). Mantener esta constitución va a depender de factores como la genética, el estilo de vida, la dieta, los hábitos alimentarios, la geografía y la edad (Novotný et al., 2019; Yatsunenko et al., 2012); por otro lado, un alteración en esta conformación [disbiosis] producirán pérdidas en las funciones normales de la microbiota y la consecuente conversión de estos microorganismos a patogénicos.
Por último, es importante recalcar que las características conformacionales de la microbiota en sus estados de eubiosis y de disbiosis son característicos de cada individuo y van a depender de los ya mencionados factores intrínsecos y extrínsecos de cada huésped. Entender estas conformaciones microbianas, van a ser trascendentales en la comprensión de los diversos procesos salud enfermedad de un individuo (Stratton et al., 2009; Wang et al., 2017)
2. Microbiota en el estado de salud
Son varios los mecanismos mediante los cuales interactúa la microbiota con el ser humano en el mantenimiento de la homeostasis; por ejemplo, a nivel metabólico, estos microorganismos aportan diversas enzimas (no codificadas por los genes humanos) que permiten una mayor obtención de energía y nutrientes de los alimentos (Turnbaugh et al., 2006), también contribuirán en la biosíntesis de vitaminas (Hill, 1997), en el señalamiento del apetito (Fetissov, 2017) y en el procesamiento xenobiótico (Das et al., 2016).
Sin embargo, quizás los hallazgos más relevantes, están enfocados al estudio de la microbiota y su relación con la actividad inmunológica, donde aparte de limitar la proliferación de patógenos oportunistas mediante la exclusión competitiva y/o la producción de sustancias antimicrobianas (Cash et al., 2006; Hooper et al., 2003), también contribuyen a la maduración, desarrollo y regulación del sistema inmunitario. Esta último punto es uno de los principales campos de estudio en la oncología, por su cercana relación con el proceso de carcinogénesis y eliminación tumoral (Bouskra et al., 2008)
3. Microbiota y enfermedad
Mecanismos moleculares como: la baja producción de inmunomoduladores, la modulación insuficiente en la proliferación de células NK y la liberación de toxinas y/o proinflamatorios (Fernández-Tomé et al., 2019; Furusawa et al., 2013; Marrero et al., 2015) son algunos de los factores patogénicos de la microbiota que pueden conducir al desarrollo de una enfermedad, especialmente en estados donde la conformación de esta se encuentra alterada o en disbiosis.
Las infecciones son una de las patologías más evidentes producto de la actividad disbiótica de la microbiota, (Amrane et al., 2019)cocktail of bacteria were proposed in order to treat Clostridium difficile infection (CDI; sin embargo, también se la ha relacionada con otras enfermedades como la obesidad (Le Chatelier et al., 2013), la diabetes tipo 2 (Qin et al., 2012), la disfunción hepática, las enfermedades autoinmunes y alergias (Ling et al., 2014) y ciertos trastornos psicológicos (Maes, 2011; Tomova et al., 2015), donde a través de mecanismo específicos, estos microorganismos contribuyen a la fisiopatología de las enfermedades descritas.
4. El cáncer: una enfermedad de gran variabilidad etiológica
El cáncer es la segunda enfermedad más letal en el mundo con un estimado de 9,9 millones de personas fallecidas en el 2020 (Cancer Today, 2020); desafortunadamente, esto se debe a la gran variabilidad etiológica, tisular y genotípica que presenta la enfermedad, lo que dificulta su diagnóstico y tratamiento (Blackadar, 2016).
La proliferación continua y no regulada de células clonales, es la principal anomalía del cáncer; esto como resultado de la acumulación de mutaciones o variantes patogénicas en regiones del ADN que se encargan del control celular (Hanahan & Weinberg, 2011), como los genes supresores de tumores, protooncogenes, genes de activación de la apoptosis y genes reparadores del ADN (Catalogue Of Somatic Mutations In Cancer, 2020; Huang et al., 2018; Stratton et al., 2009)”
Los factores ambientales también jugaran un rol importante en el proceso de carcinogénesis, especialmente la exposición a determinadas sustancias o tipos de radiación con capacidad de alterar la expresión génica de estos genes del control celular (Parsa, 2012).
El sistema inmune es otro de los pilares fundamentales en el proceso de la carcinogénesis, ya que a través de distintos mecanismos, como los mediados por los linfocitos Natural killer (NK), habrá un reconocimiento y eliminación de las células tumorales (Soloski, 2001). Las células malignas pueden evadir esta respuesta inmunológica creando condiciones hipóxicas en el microambiente tumoral, o a través de la síntesis de ciertas citoquinas (Upadhyay et al., 2018).
Aunque los factores descritos anteriormente son los más relevantes en el proceso carcinogénico, la sola interacción entre ellos no son suficientes para explicar por completo el proceso tumoral y su fisiopatología; es por ello que estudios recientes, entre otros factores, han asociado a la microbiota y su constitución como uno de los mecanismos involucrados en la aparición y progresión del cáncer (Helmink et al., 2019).
5. La microbiota en el proceso de carcinogénesis
El papel que tienen los microorganismos en la carcinogénesis es un proceso que comenzó a estudiarse desde hace más de un siglo; al inicio, estas investigaciones aportaron con datos sobre especies potencialmente oncogénicas, como: el virus del papiloma humano, Helicobacter pylori y los virus de la hepatitis B y C, entre otros (Velázquez et al., 2011), sin embargo, hoy en día proyectos como el del Microbioma Humano han demostrado que nuestra microbiota también tienen un rol en el desarrollo del cáncer (Lloyd-Price et al., 2017).
A lo largo de la última década se han descrito fuertes asociaciones epidemiológicas que relacionan el proceso neoplásico con la microbiota (Tabla1), observándose como principal característica una disminución en la cantidad y diversidad de los microorganismos en los tejidos cancerosos. También se empezaron a describir los mecanismos específicos que participan en estas interacciones y sus efectos en la promoción y eliminación del cáncer, por ejemplo del rol de la microbiota en el cáncer de pulmón con mutaciones de TP53 (Ramírez-Labrada et al., 2020), o la función de la toxina de Bacteroides fragilis en el desarrollo de cáncer colorrectal (Goodwin et al., 2011) y quizás uno de los más importantes, el influjo de los microorganismos en la respuesta inmune contra el cáncer (Erdman & Poutahidis, 2016).
Tabla 1
Algunos ejemplos de asociación epidemiológica entre la microbiota y el cáncer.
|
Tipo de cáncer |
Método de análisis |
Microorganismos involucrados y asociación |
Referencia |
|
Cáncer de mama |
Gen 16S ARNr, en tejido mamario |
Mayor abundancia relativa de Bacillus spp, Alistipes spp, Enterobacteriaceae y Staphylococcus spp. |
(Chan et al., 2016; Urbaniak et al., 2016)human breast tissue and milk were presumed to be sterile. Here, we investigated the presence of microbes in the nipple aspirate fluid (NAF |
|
Cáncer pancreático |
Gen 16S ARNr, en saliva |
Abundancia relativa de Porphyromonas gingivalis y Aggregatibacter actinomycetemcomitans (asociadas con un mayor riesgo de cáncer pacreático) y Fusobacteria spp y leptotrichia spp. |
(Fan et al., 2018; Torres et al., 2015)las relaciones directas de los microbios orales con el cáncer de páncreas no se han evaluado en estudios prospectivos. Examinamos la relación de la microbiota oral con el riesgo subsiguiente de cáncer de páncreas en un gran estudio anidado de casos y controles.\nDiseño Seleccionamos 361 adenocarcinoma incidente de páncreas y 371 controles emparejados de dos estudios de cohortes prospectivos, el Estudio II de Prevención del Cáncer de la Sociedad Americana del Cáncer y el Ensayo de detección de cáncer de próstata, pulmón, colorrectal y ovario del Instituto Nacional del Cáncer. A partir de muestras de lavado oral previas al diagnóstico, caracterizamos la composición de la microbiota oral mediante la secuenciación del gen de ARN ribosómico 16S bacteriano (ARNr 16S |
|
Cáncer de pulmón |
Gen 16S ARNr, en esputo |
Aumento relativo de Granulicatella spp, Abiotrofia spp y Streptococcus spp en pacientes con cáncer de pulmón. |
(Ran et al., 2021) |
|
Carcinoma de células escamosas de cabeza y cuello. |
Gen 16S ARNr, en saliva |
Abundancia relativa de Streptococcus spp, Dialister spp y Veillonellase spp, con disminución de Neisseria spp, Aggregatibacter spp, Haemophillus spp y Leptotrichia spp. |
(Guerrero-Preston et al., 2016)mediated by the microbiome, may be measured in saliva as head and neck squamous cell carcinoma (HNSCC |
|
Leucemia linfoblástica aguda |
Gen 16S ARNr, en heces |
Disminución en la diversidad taxonómica y abundancia relativa de Fermicutes spp, sobre el resto de filos. |
(Chua et al., 2020; Rajagopala et al., 2016)during-, and following cessation of chemotherapy.\n\nMethodology\nWe conducted a longitudinal observational study in gut microbiota profile in a group of paediatric patients diagnosed with ALL using 16 s ribosomal RNA sequencing and compared these patients’ microbiota pattern with age and ethnicity-matched healthy children. Temporal changes of gut microbiota in these patients with ALL were also examined at different time-points in relation to chemotherapy.\n\nResults\nPrior to commencement of chemotherapy, gut microbiota in children with ALL had larger inter-individual variability compared to healthy controls and was enriched with bacteria belonging to Bacteroidetes phylum and Bacteroides genus. The relative abundance of Bacteroides decreased upon commencement of chemotherapy. Restitution of gut microbiota composition to resemble that of healthy controls occurred after cessation of chemotherapy. However, the microbiota composition (beta diversity |
|
Cáncer colorrectal |
Gen 16S ARNr, en heces |
Enriquecimiento significativo de Porphyromonas spp, Fusobacterium spp y Parvimonas spp en cáncer colorecatal. |
(Mo et al., 2020) |
El principal hallazgo en el estudio de las microbiotas en el cáncer es la disminución relativa de ciertas especies y el enriquecimiento de otras, teniendo estas alteraciones un efecto directo sobre la promoción y eliminación de los tumores.
Sin embargo, el Consorcio Internacional del Microbioma Humano en el 2019, concluyó que no existe una evidencia directa que indique que los microorganismos que nos colonizan, sean una determinante clave en la etiopatogenia del cáncer; Scott et al. afirma que la microbiota en conjunto con los factores ambientales y las características tanto genéticas como epigenéticas del huésped, representan un vértice de interacción molecular que impulsa y/o media la carcinogénesis (A. J. Scott et al., 2019).
De manera general y basándonos en el marco conceptual de lo que es un carcinógeno (R. E. Scott et al., 1984), describiremos algunos mecanismo moleculares [integración genómica, genotoxicidad, inflamación, inmunidad y metabolismo] (Schwabe & Jobin, 2013), por los cuales la microbiota podría iniciar el proceso de carcinogénesis:
Integración genómica: es un evento molecular importante observado en la carcinogénesis de ciertos tipos de cánceres, entre ellos el urogenital, el de cabeza y el de cuello. En este mecanismo los microorganismos acoplan sus genes a los del huésped (Narisawa-Saito & Kiyono, 2007), distribuyéndose las inserciones de manera aleatoria por todo el genoma con una marcada predisposición por los sitios genómicos frágiles (Fernández-Tomé et al., 2019). Resultado de este acoplamiento, puede haber un aumento en la expresión de proteínas con efectos carcinogénicos o a su vez una desregulación de los genes supresores de tumores; éste mecanismo se asocia frecuentemente con los virus (Cook et al., 2017).
Genotoxicidad: ciertas cepas microbianas tienen la capacidad de sintetizar toxinas y productos metabólicos con efectos negativos para la estructura del ADN del huésped. Las variantes patológicas provocadas por este mecanismo incluyen roturas de hebras, deleciones y reordenamientos (Attene-Ramos et al., 2006; He et al., 2019). Estas alteraciones, si no terminan con la muerte celular, pueden subsistir y afectar a genes encargados de la regulación de la proliferación celular, conllevando al posible desarrollo de neoplasias. Como ejemplo de este mecanismo están las toxinas distensora citoletal y colibactina de ciertas Enterobacteriaceae (Buc et al., 2013).
Inflamación: elementos virulentos microbianos son capaces de inducir en el huésped estados crónicos de inflamación tanto dentro de los nichos microbianos como fuera de ellos (órganos distales). El mecanismo molecular se origina por la interacción de los microorganismos con los receptores de tipo toll (TLR) (Buchta Rosean & Rutkowski, 2017), produciendo alteraciones que puede desembocar en una proliferación celular desregulada y/o en el fracaso de la apoptosis (Francescone et al., 2014; Wu et al., 2007)
Inmunidad: la microbiota tiene un papel importante en procesos que van desde la promoción de la inmunovigilancia cancerígena, a través de la mejora en la intensidad de las respuestas inmunitarias y la ampliación en el número de receptores de las células T (Zitvogel et al., 2016), hasta la neutralización de la citotoxicidad antitumoral (Kostic et al., 2013).
Metabolismo: el metabolismo microbiano, en especial el bacteriano-intestinal (por ser el más abundante y por las diversas vías metabólicas que aporta), está estrechamente relacionado en la prevención del proceso de carcinogénesis (Lloyd-Price et al., 2017). Actualmente, se está estudiando el posible rol que tiene la fermentación bacteriana de la fibra dietética a ácidos grasos de cadena corta (butirato) en la supresión de la oncogénesis del cáncer colorrectal, esto debido talvez a los efectos antiinflamatorios y antiproliferativos que tienen estos productos metabólicos (Ríos-Covián et al., 2016). Por el contrario, el metabolismo de ácidos biliares y algunas proteínas, produce aminas aromáticas y sulfuros que tienen un alto potencial cancerígeno (O’Keefe et al., 2015).
Estos mecanismos carcinogénicos, pueden ser propiedades inherentes de un género microbiano en especial, o resultado del proceso disbiótico de la microbiota en su conjunto en un nicho determinado. Para explicar el proceso de carcinogénesis mediado por microorganismos se ha planteado dos teoría que se complementan entre sí, la primera hace referencia a la existencia de un “Patógeno alfa”, el cual inicia el proceso neoplásico [p.ej: Bacteroides fragilis y la toxina enterotoxigénica en el cáncer colorrectal] (Sears & Pardoll, 2011). Sin embargo, esta teoría no explica ¿por qué? cuando se examina las microbiota en pacientes con cáncer, no siempre se identifica un patógeno potencialmente cancerígeno (Kinross et al., 2017; Marchesi et al., 2011). Entonces, como un refinamiento a esta teoría, se ha propuesto el modelo “conductor-pasajero”, en el cual, igualmente se requiere la actuación de un microorganismo clave pero luego, debido a los cambios sufridos en el microambiente, la microbiota se transforma de beneficiosa a una potencialmente cancerígena con proliferación de microorganismos oportunistas (Tjalsma et al., 2012). [Figura 1].
Figura 1
Mecanismos de la microbiota en la carcinogénesis
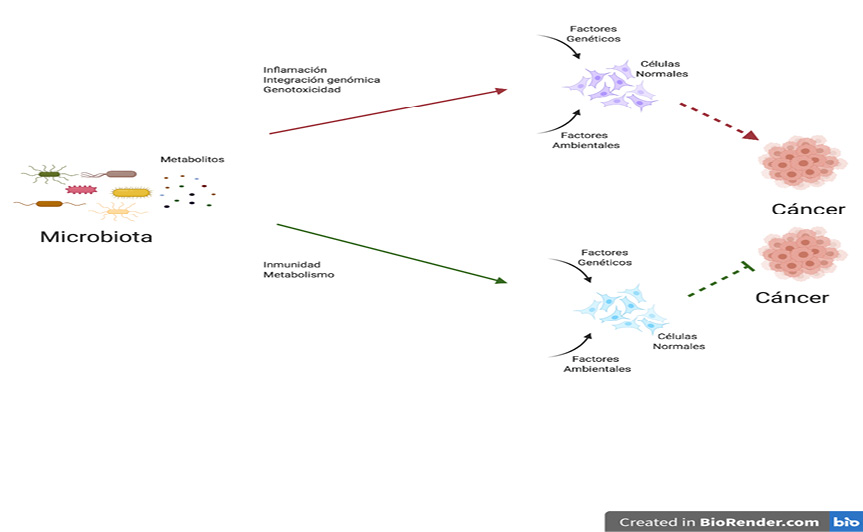
Existen dos posibles vías los cuales pueden inhibir o favorecer la formación de tumores.
6. Efectos antitumorales de la microbiota en la oncogénesis
El principal mecanismo de nuestro organismo para hacer frente al crecimiento de células malignas en estadios tempranos, es la inmunovigilancia tumoral (Dunn et al., 2004), la cual se encuentra conformada por las células T-CD4 secretoras de interleucina 9 (IL-9); esto es importante debido a que la secreción de estas citocinas permitirá un mayor reclutamiento de células dendríticas, mastocitos, células NK y células T-CD8 en el microambiente tumoral (Lu & Yi, 2013; Végran et al., 2015), con capacidad de eliminará el tumor y mejorará la supervivencia del paciente con cáncer (Lu et al., 2012; Lu & Yi, 2013).
Se ha demostrado que nuestra microbiota, especialmente la intestinal participa también de este mecanismo, a través de la secreción de factor de crecimiento transformante beta (TGF-β) y la interleucina 4 (IL-4), que igualmente estimularán la secreción de IL-9 por parte de las células T (Apetoh, 2020; Schmitt et al., 1994).
También un experimento realizado por Almeida et al. (2020), describe como ratones libres de gérmenes o con tratamiento antibiótico de amplio espectro, presentan una menor expresión de TGF-β y IL-4 en relación a ratones convencionales, dando como resultado una disminución en la frecuencia de células T-CD4 productoras de IL-9 produciendo en estos animales, una mayor susceptibilidad en el desarrollo de tumores.
7. Microbiota y su relación en ciertos tipos de cáncer
7.1. Cáncer gastrointestinal
Nuestro intestino esta colonizado por alrededor de 3,8 x 1013 bacterias (Sender et al., 2016) con una diversidad taxonómica relativamente estable (Qin et al., 2010), sin embargo, en el cáncer colorrectal, puede truncarse esta conformación, favoreciendo la proliferación de ciertos taxones microbianos como de las familias Fusobacteriaceae y Streptococcaceae o especies de los géneros Bacteroides spp. y Parabacteroides spp (Feng et al., 2015; Kostic et al., 2012) con distintos efectos sobre el crecimiento celular de estas células patológicas (Taddese et al., 2020.)
A nivel gástrico, el principal agente microbiano cancerígeno es el Helicobacter pylori que mediante la secreción de oncoproteínas y toxinas, estimula la respuesta inmune hacia un entorno inflamatorio. Además, altera las vías de señalización de la secreción de ácido clorhídrico que conducen a la aclorhidria, atrofiando de este modo el epitelio gastrointestinal y provocando la displasia (Dadashzadeh et al., 2017). Hoy en día, las tecnologías de secuenciación genética nos han permitido identificar otros microrganismos que cooperan con esta infección (Maldonado-Contreras et al., 2011), y el rol de otras bacterias en los casos de cáncer gástrico negativo para la presencia de Helicobacter pylori (Guo et al., 2020)
Los estudios acerca de los mecanismos moleculares, a través de los cuales las especies microbianas de la microbiota gastrointestinal, promueven o interfieren en el desarrollo del cáncer son aún muy limitados, pero cada vez se identifican nuevos microorganismos y sus metabolitos relacionados con el cáncer, por ejemplo Campylobacter jejuni y la toxina de distensión citoletal en el cáncer colorrectal (He et al., 2019), o ciertas cepas de Escherichia coli productoras de cilomodulina y genotoxina en el cáncer de colon (Buc et al., 2013). También las especies de la microbiota colónica normal pueden inducir al desarrollo del cáncer, si existen factores predisponentes en el huésped (Attene-Ramos et al., 2006).
7.2. Cáncer de páncreas
El adenocarcinoma ductal pancreático es una neoplasia con una altísima mortalidad, siendo su supervivencia general a 5 años del 9% (Siegel et al., 2020), incluso con la resección quirúrgica, la tasa de recurrencia es muy alta. Sin embargo, existe un porcentaje mucho menor de pacientes, sin diferencias genómicas importantes en sus cánceres, que después de la cirugía sobreviven más de 5 años (Molin et al., 2015). Utilizando métodos de secuenciación, se ha identificado que los pacientes con larga supervivencia, tienen una constitución microbiana intestinal diferente a los pacientes de corta supervivencia, y que la firma microbiana de los pacientes con larga supervivencia, está conformada por Pseudoxanthomonas, Streptomyces, Saccharopolyspora, Bacillus clausii siendo un posible factor positivo en la respuesta inmune contra este tumor (Riquelme et al., 2019).
7.3. Cáncer de pulmón
La etiología del cáncer de pulmón ha sido asociada a la inflamación crónica, sin embargo, los mediadores inmunitarios para que se produzca el mismo, no han sido del todo dilucidados. Existe cada vez más evidencia, que relaciona la microbiota local de la mucosa del árbol respiratorio con el desarrollo de adenocarcinoma pulmonar, debido a la activación de células T gamma delta (γδ) residentes en el pulmón. En estudios experimentales, con ratones con una mucosa libre de gérmenes, o aquellos que fueron tratados con antibioticoterpia, se demostró un significativo factor protector para el desarrollo de cáncer de pulmón inducido por mutaciones K-ras y p53. Todo esto se va a traducir en la formación de citoquinas, quimioquinas y prostaglandinas que estimulan la proliferación celular, angiogénesis, remodelación tisular y metástasis en el pulmón.
Los propios gérmenes comensales de la microbiota pulmonar pueden ser, por ellos mismos, un factor promotor para el desarrollo del cáncer, sin embargo, los microorganismos considerados no comensales son los que van a iniciar el desarrollo de un tumor según varios estudios. Los géneros bacterianos que más comúnmente se encuentran en la microbiota pulmonar son Staphylococcus spp (15%), Streptococcus spp (15%), Lactobacillus spp (15%), así como miembros de la familia Pasteurellaceae. Sin embargo, cuando hay un desbalance tanto en las proporciones como en la cantidad absoluta de estos gérmenes, pueden actuar como un factor determinante para el desarrollo de tumores (Jin et al., 2019).
8. Virioma en el cáncer
En el año 1911, Peyton Rous investigador de la Universidad Johns Hopkins, descubrió que un virus podría ser el agente causal de un tumor maligno (Rous, 1911), poco después se describieron los mecanismos genéticos involucrados en este proceso, y para 1964, se había aisló el primer virus capaz de inducir cáncer en humanos, se trataba del virus de Epstein-Barr y el linfoma de Burkitt. (Epstein et al., 1964). Sin embargo la investigación acerca de este tema muchas veces ha estado limitada, debido sobre todo a las dificultades de aislamiento y caracterización de estos microorganismos.
Nuestro virioma está compuesto por una gran diversidad de virus ARN y ADN que colonizan nuestro intestino, piel y cavidad bucal, ya sea de manera transitoria o crónica, con capacidad de infectar tanto a células como bacterias (Abeles et al., 2014; Hannigan et al., 2015).
Los mecanismos a través de los cuales nuestro virioma pueden inducir la carcinogénesis incluyen: alteraciones genéticas, efectos mecanicistas sobre las vías metabólicas celulares, inflamación crónica y destrucción de la comunidad bacteriana eubiótica por bacteriófagos; este último mecanismo muy importante en los cánceres gastrointestinales, donde por la lisis bacteriana, pueden haber una promoción de la carcinogénesis al permitir la proliferación de bacterias oportunistas como Fusobacterium nucleatum (Kostic et al., 2013).
En cuento a la prevención del cáncer, nuestro virioma, específicamente el conformado por los bacteriófagos contribuyen a la prevención del mismo, ya que pueden ir dirigidos contra bacterias potencialmente patógenas, infectándolas y destruyéndolas (Abedon et al., 2011).
Finalmente se ha planteado el uso de nuestro virioma como método de diagnóstico y pronóstico de ciertos cánceres. Por ejemplo se está buscando marcadores taxonómicos virales asociados a algunos cánceres gastrointestinales en muestras fecales mediante el análisis metagenómico de escopeta (Nakatsu et al., 2018), y hasta el momento los estudios han tenido buenos resultados, logrando identificar ciertas firmas en el virioma que se asociarían a determinados tipos de cánceres (Hannigan et al., 2018).
9. La microbiota y su efecto en la terapia antitumoral
Desde hace varios años se ha hecho revisiones sobre este tema, pero es quizás en los últimos dos años, cuando más se ha investigado al respecto. La interacción entre la microbiota humana y las drogas anticancerosas fue acuñado bajo el término de fármaco-microbiómica por ElRakaiby en 2014 (ElRakaiby et al., 2014). El impacto dual de la microbiota humana para alterar los agentes quimioterapéuticos, y viceversa se convirtió en un dilema filosófico: “estos es una causa o un efecto”.
Los mecanismos por los cuales la microbiota intestinal altera los objetivos de la terapia oncológica pueden resumirse en: translocación, inmunomodulación, metabolismo, degradación enzimática, y diversidad bacteriana reducida (Hekmatshoar et al., 2019).
10. Quimioterapia
La interacción directa con las bacterias puede afectar la eficacia de los fármacos quimioterapéuticos. De alrededor de 30 drogas probadas in vitro, en la presencia de microorganismos no patógenos Gram negativos (Escherichia coli) y Gram positivos (Listeria welshimeri), 10 de ellos han sido inhibidos con eficacia por una o ambas especies, mientras que en 6 de ellos la actividad ha sido potenciada (Roy & Trinchieri, 2017).
La microbiota intestinal parece tener un rol fundamental en 4 aspectos: 1) carcinogénesis gastrointestinal 2) modulación de la actividad y toxicidad de muchos agentes de quimioterapia e inmunoterapia 3) mediante la manipulación de la misma se puede mejorar la eficacia de los agentes antitumorales 4) la toxicidad de varios fármacos para el tratamiento del cáncer puede disminuirse mediante la manipulación de la microbiota (Gori et al., 2019).
Para referirnos a algunos ejemplos de cómo puede haber una interacción microbiota-antitumorales-eficacia-toxicidad, citamos que los antibióticos disminuyen la acción de uno de los quimioterápicos más comunes como lo es el 5-Fluoracilo (5FU), y cuando hay un desbalance entre los comensales normales del intestino, con un incremento de Escherichia coli y Enterococcus spp, la toxicidad de este fármaco aumenta, pudiendo llegar a producir mucositis, bacteremia y sepsis.
Los antimicrobianos contra Gram positivos, disminuyen la eficacia de las sales de platino utilizados en la terapia contra el cáncer epitelial de varias localizaciones del cuerpo. La ingesta del aminoácido metionina en la dieta, tiene un efecto protector para la toxicidad del Cisplatino, al promover que proliferen bacterias beneficiosas como Lachnospiraceae spp. y Lactobacillus spp. (Campbell et al., 1996)
Para la Gemcitabina el tratamiento con antibióticos tiene el efecto opuesto, puede potenciar la eficacia de este fármaco (Choy et al., 2018). Para evaluar la hipótesis de la resistencia a Gemcitabina causadas por bacterias intratumorales, se analizó tejido pancreático tumoral, encontrando ADN bacteriano en 86 de 113 pacientes (76%), vs 3 de 120 (15%) de ADN bacteriano encontrado en tejido pancreático normal obtenido de donantes de órganos. Este y una serie de estudios más nos llevan a pensar que la microbiota intratumoral puede ser en el futuro un blanco terapéutico dentro de la extensa gama de terapia anticancerosa (Geller et al., 2017).
La inhibición de la Beta glucoronidasa bacteriana producida por Escherichia coli, reduce la incidencia de diarrea inducida por Irinotecan, un inhibidor de la topoisomerasa utilizado en cáncer avanzado de colon, estómago, páncreas y cáncer de pulmón de células pequeñas.
Una mención singular constituye, la descripción de cómo la microbiota de los tumores de la mama puede variar en función de la quimioterapia neoadyuvante administrada, e influye en la evolución natural de la enfermedad. Los microorganismos presentes en los tumores mamarios son distintos a los del resto de la glándula mamaria. Se ha visto que en la microbiota tumoral de las pacientes que reciben quimioterapia neoadyuvante hay una elevación y sobreexpresión de Pseudomonas spp y disminución de Prevotella spp Así mismo en aquellas pacientes que presentaron actividad metástasis se vio un incremento en las poblaciones de Brevundimomas spp y Staphylococcus spp en la microbiota tumoral (Chiba et al., 2020).
11. Inmunoterapia
La inmunoterapia en los últimos años ha tenido un gran avance, sobre todo los anticuerpos monoclonales que actúan bloqueando el antígeno 4 de los Linfocitos T citotóxicos (CTLA-4) (Leach et al., 1996), o aquellos que actúan en la proteína de muerte celular programada (PD-1 Y PD-L1). Cabe recalcar, que se ha evidenciado que no todos los pacientes tienen una respuesta óptima al tratamiento, algunos por ejemplo, terminan en regresiones tumorales dramáticas y en otros el tratamiento no les presentó ningún beneficio (Vétizou et al., 2015). Múltiples estudios han demostrado que la microbiota intestinal afecta la eficacia terapéutica de estos inhibidores de control inmune. La mucosa intestinal comprende una capa única de células epiteliales linfocitos intraepiteliales, por lo que hay una interacción directa con el sistema inmune. Diversos estudios han demostrado que especies de Bifidobacterium spp se incrementan en la microbiota, favoreciendo el efecto terapéutico de anti PD1/PDL1 (nivolumab, atezolizumab), con un lento crecimiento tumoral. En estudios experimentales la administración oral de Bacteroides fragilis y cepas de Burkholderia spp, mejoran la eficacia de la terapia anti CTLA-4 (ipilimumab) (W. Li et al., 2019; Vétizou et al., 2015).
En la inmunoterapia con anti PD-1/PD-L1, la sobrevida global y la sobrevida libre de progresión es significativamente mayor en pacientes con tumores epiteliales que no recibieron tratamiento antibiótico de rutina, si se los compara con aquellos que si recibieron tratamiento antibiótico. Así mismo la toxicidad de esta terapia inmunológica se puede ver afectada por la microbiota, especialmente para la aparición de colitis (Routy et al., 2018).
12. Radioterapia
La radioterapia no produce únicamente cambios en el ADN de las células blanco, sino también produce cambios en células cercanas por fenómenos de inflamación, reactividad inmunitaria e inestabilidad genómica. Se cree que al igual como sucede con la quimioterapia y la inmunoterapia, la microbiota intestinal tiene un efecto en la respuesta inmunitaria inducida por las células muertas inmunogénicas. Así mismo esto afecta a la toxicidad, ya que los cambios observados, secundarios a la radioterapia en la composición de la microbiota de las superficies epiteliales, contribuye a la patogénesis de las mucositis orales, diarrea, enteritis, colitis y fallo de la médula ósea (Barker et al., 2015).
13. Conclusiones
El empleo de las nuevas herramientas investigativas, sobretodo la relacionadas a la genómica, nos han permitido ahondar en las funciones que desempeñan los microrganismos que colonizan nuestro cuerpo en el proceso salud enfermedad sin embargo, aún queda mucho por hacer, por ejemplo está el desafío de identificar especies microbianas individuales y sus efectos.
Sería muy importante analizar la capacidad de ciertos microorganismos para alterar la estructura del ADN y específicamente conocer si poseen sitios selectivos para modificar ciertos genes involucrados en la supresión tumoral o cuales serían los elementos que favorecen están modificaciones. Cabe recordar que la información actual no permite confirmar si la microbiota favorece o inhibe la formación tumoral pero sugiere un posible rol en los mismos, dependiendo del ambiente tumoral, del sistema inmunológico y sobretodo del tipo de tumor.
Existen ciertas localizaciones tumorales, en las cuales se ha estudiado más la influencia de la microbiota, entre los que resaltan el cáncer gastrointestinal, cáncer de páncreas, cáncer de pulmón y cáncer de mama, sin embargo la mayor parte de neoplasias tienen su propia microbiota que las caracteriza. Existen microorganismos que se han descrito desde hace décadas en la etiopatogenia de las enfermedades neoplásicas como el Helicobacter pylori o el virus de Epstein-Barr, sin embargo hay un gran número de otras bacterias y virus, implicadas tanto como factor protector como factor causal de varios tipos de cáncer. Los distintos elementos de la microbiota pueden influir de manera importante en la terapia antitumoral, ya sea potenciando o disminuyendo los efectos de la misma. Los principales tipos de modalidades terapéuticas que se ven afectados por el efecto de la microbiota son: la quimioterapia, terapia biológica, inmunoterapia y la radioterapia. La capacidad de que en un futuro podamos identificar un blanco tumoral, con epicentro en la microbiota de un tumor es sumamente alentador, con el fin de tener cada vez mayores y mejores armas en el área de la oncología.
Agradecimientos
La figura incluida fue creada con BioRender.com.